在OpenCL兼容GPU上进行对象分类#
APOC基于pyclesperanto和scikit-learn。它允许根据测量的属性/特征(如强度、形状和相邻细胞数量)对对象进行分类。
import apoc
from skimage.io import imread, imsave
import pyclesperanto_prototype as cle
import numpy as np
import matplotlib.pyplot as plt
对于对象分类,我们需要一个强度图像和一个标签图像作为输入。
# load intensity image
image = imread('../../data/blobs.tif')
# segment the image
labels = cle.label(cle.threshold_otsu(image))
fig, axs = plt.subplots(1, 2)
cle.imshow(image, color_map="Greys_r", plot=axs[0])
cle.imshow(labels, labels=True, plot=axs[1])

训练#
我们还需要一个地面真实标注图像。这个图像也是一个具有稀疏标注的标签图像。通过所有应该属于类别1的对象画了一条值为1的线。通过所有应该被分类为类别2的对象画了一条值为2的线。如果线穿过背景,这将被忽略。在这个例子中,对象被标注为三个类别:
细长的对象
圆形对象
小对象
annotation = cle.push(imread('../../data/label_annotation.tif'))
fig, axs = plt.subplots(1, 2)
cle.imshow(labels, labels=True, plot=axs[0])
cle.imshow(annotation, labels=True, plot=axs[1])
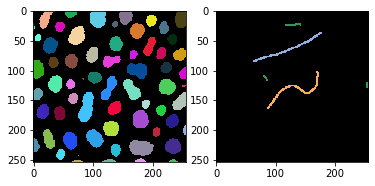
接下来,我们需要定义我们想要用于分类对象的特征。我们将使用面积、形状和强度的标准差。
features = 'area mean_max_distance_to_centroid_ratio standard_deviation_intensity'
# Create an object classifier
filename = "../../data/blobs_object_classifier.cl"
classifier = apoc.ObjectClassifier(filename)
# train it; after training, it will be saved to the file specified above
classifier.train(features, labels, annotation, image)
在分类器训练完成后,我们可以立即使用它来预测图像中对象的分类。
# determine object classification
classification_result = classifier.predict(labels, image)
cle.imshow(classification_result, labels=True)

预测#
你也可以从磁盘重新加载分类器并将其应用于其他图像。我们将通过旋转原始图像来模拟这一过程。顺便说一下,这是一个很好的健全性检查,可以看看分类是否依赖于图像的方向。
image2 = cle.rotate(image, angle_around_z_in_degrees=90)
labels2 = cle.rotate(labels, angle_around_z_in_degrees=90)
classifier2 = apoc.ObjectClassifier("../../data/blobs_object_classifier.cl")
classification_result2 = classifier2.predict(labels2, image2)
cle.imshow(classification_result2, labels=True)

可用于对象分类的特征#
我们可以打印出所有可用的特征。带有?的参数在该位置期望一个数字,并且可以多次指定多个值。
apoc.list_available_object_classification_features()
['label',
'original_label',
'bbox_min_x',
'bbox_min_y',
'bbox_min_z',
'bbox_max_x',
'bbox_max_y',
'bbox_max_z',
'bbox_width',
'bbox_height',
'bbox_depth',
'min_intensity',
'max_intensity',
'sum_intensity',
'area',
'mean_intensity',
'sum_intensity_times_x',
'mass_center_x',
'sum_intensity_times_y',
'mass_center_y',
'sum_intensity_times_z',
'mass_center_z',
'sum_x',
'centroid_x',
'sum_y',
'centroid_y',
'sum_z',
'centroid_z',
'sum_distance_to_centroid',
'mean_distance_to_centroid',
'sum_distance_to_mass_center',
'mean_distance_to_mass_center',
'standard_deviation_intensity',
'max_distance_to_centroid',
'max_distance_to_mass_center',
'mean_max_distance_to_centroid_ratio',
'mean_max_distance_to_mass_center_ratio',
'touching_neighbor_count',
'average_distance_of_touching_neighbors',
'average_distance_of_n_nearest_neighbors=?',
'average_distance_of_n_nearest_neighbors=?']
