标签边缘#
在处理图像中的生物对象(如细胞和细胞核)时,识别位于对象表面的所有像素可能是有意义的。 本笔记本演示了如何选择细胞核边界上的像素,以防我们想要测量核膜中的强度。
import pyclesperanto_prototype as cle
import numpy as np
from skimage.io import imread
image = cle.asarray(imread("../../data/mitosis_mod.tif")[0:40,25:65])
image
|
|
cle._ image
|
然后我们对细胞核进行分割。
label_image = cle.voronoi_otsu_labeling(image, spot_sigma=2, outline_sigma=1)
label_image
|
|
cle._ image
|
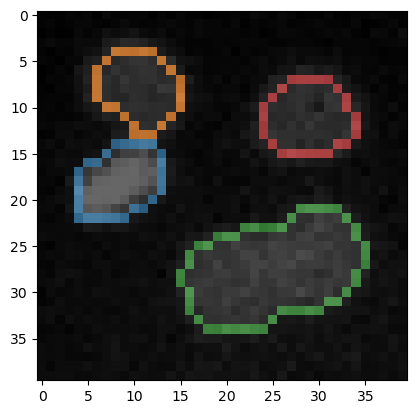
从细胞核标签图像中,我们可以提取另一个标签图像,其中包含位于标签边缘的所有像素。
edge_label_image = cle.reduce_labels_to_label_edges(label_image)
edge_label_image
|
|
cle._ image
|
如果想要在边界周围测量更厚的区域,我们可以扩展边界。
thicker_edges = cle.dilate_labels(edge_label_image, radius=1)
thicker_edges
|
|
cle._ image
|
为了可视化目的,我们还可以查看带有标签边界的原始图像。
cle.imshow(image, continue_drawing=True)
cle.imshow(edge_label_image, alpha=0.6, labels=True)