高斯-大津-标记#
到目前为止,我们已经使用了诸如高斯模糊等滤波器、大津法等阈值算法,并使用连通组件标记来标记对象。这三种算法的组合特别常见,以至于有便捷函数可以一次性执行这三个操作。这种图像分割工作流是一种非常简单的方法,例如用于检测和分割荧光显微镜图像中的细胞核。如果对象不太密集,这种方法会很有效。
from skimage.io import imread
import matplotlib.pyplot as plt
import pyclesperanto_prototype as cle
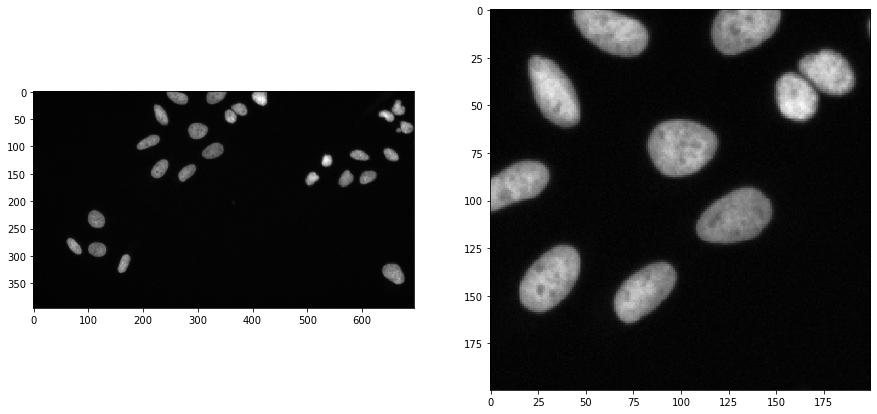
为了演示这个工作流程,我们使用来自Broad生物图像挑战的图像数据: 我们使用了图像集BBBC022v1 Gustafsdottir et al., PLOS ONE, 2013,该数据集可从Broad生物图像基准集合获得 Ljosa et al., Nature Methods, 2012。
我们加载图像,并出于可视化目的,裁剪出一个子区域:
input_image = imread("../../data/BBBC022/IXMtest_A02_s9.tif")[:,:,0]
input_crop = input_image[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(input_image, plot=axs[0])
cle.imshow(input_crop, plot=axs[1])
应用算法#
高斯-大津-标记是pyclesperanto和可编写脚本的napari插件napari-segment-blobs-and-things-with-membranes中的一个命令。该操作有一个outline_sigma参数,用于控制分割对象轮廓的精确度。
sigma_outline = 1
segmented = cle.gauss_otsu_labeling(input_image, outline_sigma=sigma_outline)
segmented_crop = segmented[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(segmented, labels=True, plot=axs[0])
cle.imshow(segmented_crop, labels=True, plot=axs[1])

sigma_outline = 7
segmented = cle.gauss_otsu_labeling(input_image, outline_sigma=sigma_outline)
segmented_crop = segmented[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(segmented, labels=True, plot=axs[0])
cle.imshow(segmented_crop, labels=True, plot=axs[1])

如你所见,如果对象太靠近,它们将被一起分割。在这些情况下,建议尝试Voronoi-大津-标记。
