多维图像堆栈#
多维图像数据可以以类似于多通道图像数据的方式处理。
三维图像堆栈#
还有具有三个空间维度的图像:X、Y和Z。在显微镜和医学成像中可以找到典型的例子。让我们来看一个磁共振成像(MRI)数据集:
from skimage.io import imread
from pyclesperanto_prototype import imshow
image_stack = imread('../../data/Haase_MRT_tfl3d1.tif')
image_stack.shape
(192, 256, 256)
我们看到数据确实有三个维度,在这种情况下是192个Z平面和256个X和Y像素。我们可以使用pyclesperanto的imshow来显示它:
imshow(image_stack)
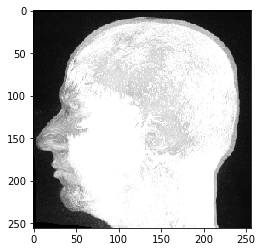
这个MRI数据集看起来不寻常,因为我们看到的是最大强度投影,这是pyclesperanto默认的三维数据可视化方式。
图像切片#
我们可以通过指定3D numpy数组中的索引来检查单个图像切片,这次我们使用Matplotlib的imshow进行可视化:
import matplotlib.pyplot as plt
fig, axs = plt.subplots(1, 3, figsize=(15,15))
# show three planar images
axs[0].imshow(image_stack[48], cmap='Greys_r')
axs[1].imshow(image_stack[96], cmap='Greys_r')
axs[2].imshow(image_stack[144], cmap='Greys_r');
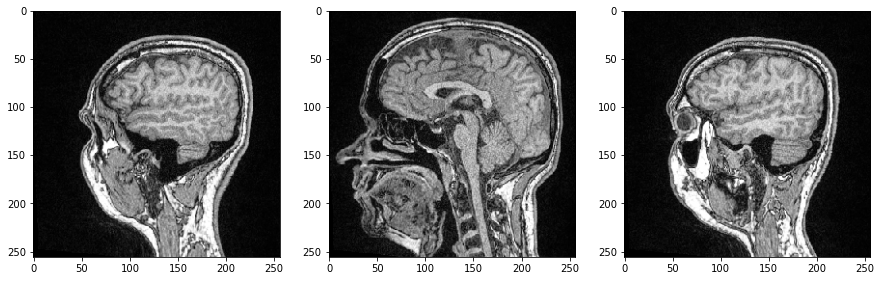
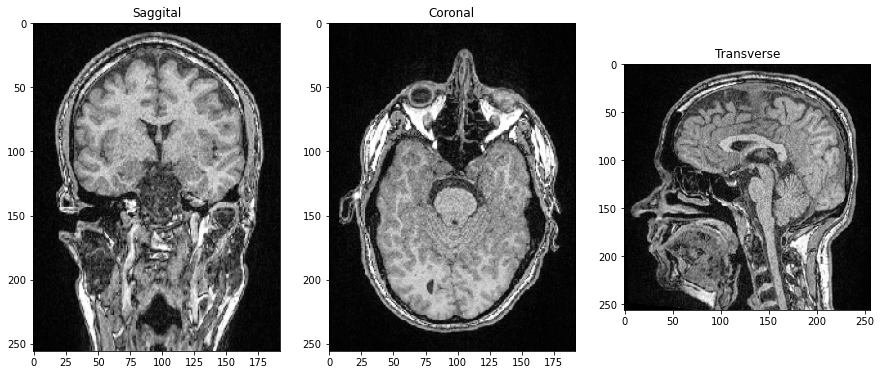
由于所有三个维度都是空间维度,我们还可以制作垂直于图像平面的切片,对应于解剖平面。为了正确定向图像,我们可以通过在末尾添加.T来转置它们的轴。
saggital = image_stack[:,:,128].T
coronal = image_stack[:,128,:].T
transverse = image_stack[96]
fig, axs = plt.subplots(1, 3, figsize=(15,15))
# show orthogonal planes
axs[0].imshow(saggital, cmap='Greys_r')
axs[0].set_title('矢状面')
axs[1].imshow(coronal, cmap='Greys_r')
axs[1].set_title('冠状面')
axs[2].imshow(transverse, cmap='Greys_r')
axs[2].set_title('横断面');
视频#
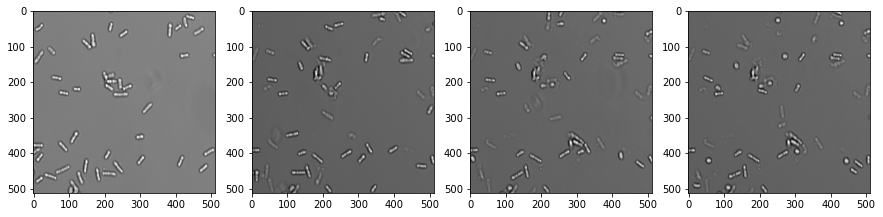
如果图像数据集有时间维度,我们称之为视频。处理视频的方式类似于多通道图像和图像堆栈。让我们打开一个显示酵母细胞随时间变圆的显微镜数据集。(图像数据由Anne Esslinger,Alberti实验室,MPI CBG提供)
video = imread('../../data/rounding_assay.tif')
video.shape
(64, 512, 512)
fig, axs = plt.subplots(1, 4, figsize=(15,15))
# show three planar images
axs[0].imshow(video[0], cmap='Greys_r')
axs[1].imshow(video[5], cmap='Greys_r')
axs[2].imshow(video[10], cmap='Greys_r')
axs[3].imshow(video[15], cmap='Greys_r');
n维数据#
高维数据在显微镜领域很常见。为了正确处理它们,必须仔细研究图像数据集具有哪些维度。我们可以使用mitosis数据集探索可能性:
mitosis = imread('../../data/mitosis.tif')
mitosis.shape
(51, 5, 2, 196, 171)
提示:在ImageJ/Fiji中打开数据集以了解这些数字代表什么。你可以看到有细胞分裂数据集具有
51帧,
5个Z切片,
2个通道 和
171 x 196像素大小。
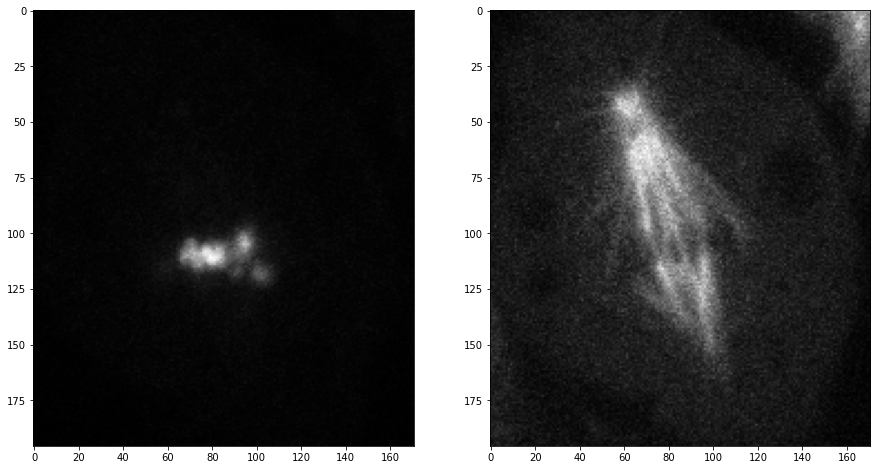
我们现在获取第一个时间点(索引0)中心平面(索引2)的通道1和2:
timepoint = 0
plane = 2
channel1 = mitosis[timepoint, plane, 0]
channel2 = mitosis[timepoint, plane, 1]
fig, axs = plt.subplots(1, 2, figsize=(15,15))
axs[0].imshow(channel1, cmap='Greys_r')
axs[1].imshow(channel2, cmap='Greys_r');
练习#
打开细胞分裂数据集,选择三个时间点并并排显示它们。结果图应该有三列两行。在第一行显示channel1,第二行显示channel2。
