术语#
这个笔记本用于展示不同种类的图像和分割图像,以解释常用术语。
from skimage.io import imread, imshow
import napari_segment_blobs_and_things_with_membranes as nsbatwm
import stackview
import numpy as np
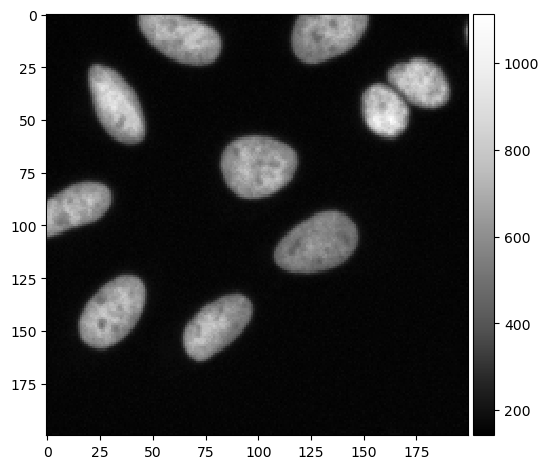
强度图像#
我们从显微镜常常产生的强度图像开始。
input_image = imread("../../data/BBBC022/IXMtest_A02_s9.tif")[:,:,0]
cropped_image = input_image[0:200, 200:400]
imshow(cropped_image, cmap='Greys_r')
C:\Users\haase\mambaforge\envs\bio39\lib\site-packages\skimage\io\_plugins\matplotlib_plugin.py:150: UserWarning: Low image data range; displaying image with stretched contrast.
lo, hi, cmap = _get_display_range(image)
<matplotlib.image.AxesImage at 0x2ba68647e20>
二值图像#
基本的分割算法会产生二值图像。它们的特点是只有两种不同的强度,如0和1。
binary_image = nsbatwm.threshold_otsu(cropped_image)
binary_image
|
|
nsbatwm made image
|
实例分割#
如果每个单独的对象都有自己的强度/颜色,我们称之为实例分割标签图像。实例用整数标记。这个图像中的最大强度(标签)通常对应于对象的数量。
instance_segmentation = nsbatwm.voronoi_otsu_labeling(cropped_image,
spot_sigma=5,
outline_sigma=1)
instance_segmentation
|
|
nsbatwm made image
|
当你在本地运行这个笔记本时,你可以用鼠标悬停在图像上并检查像素强度。
import stackview
stackview.picker(instance_segmentation)
语义分割#
语义分割标签图像可以有两个以上的标签,通常标记具有相同含义的像素区域,例如:细胞核、核膜和背景。
semantic_segmentation = binary_image + nsbatwm.maximum_filter(binary_image).astype(np.uint32) + 1
semantic_segmentation
|
|
nsbatwm made made image
|
稀疏标注#
标注的标签图像通常由人工绘制。在本地执行这个笔记本时,你可以绘制一些标注。如果你精确地标注几个细胞核,你就创建了一个稀疏的细胞核标注。按住ALT键可以擦除标注。
sparse_label_annotation = np.zeros_like(cropped_image, dtype=np.uint32)
stackview.annotate(cropped_image, sparse_label_annotation)
stackview.insight(sparse_label_annotation)
|
|
|
你也可以创建一个稀疏的语义标注,例如,通过标注细胞核内的一些像素和背景中的一些像素。
sparse_semantic_annotation = np.zeros_like(cropped_image, dtype=np.uint32)
stackview.annotate(cropped_image, sparse_semantic_annotation)
stackview.insight(sparse_semantic_annotation)
|
|
|
