图像二值化#
在对图像进行二值化时,我们生成的图像只有两个值:真和假。它们也可能包含值0,例如用于背景,而其他任何值用于前景。
import numpy as np
from skimage.io import imread, imshow
from skimage.filters import gaussian
我们使用这个细胞核的示例图像。
image_nuclei = imread('../../data/mitosis_mod.tif')
imshow(image_nuclei)
<matplotlib.image.AxesImage at 0x1dcf6b5dbe0>

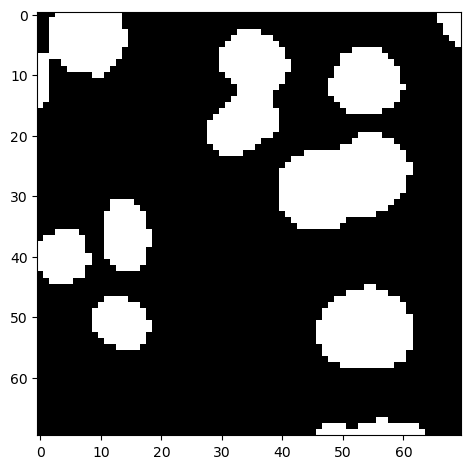
图像阈值化#
最常见的二值化技术是阈值化。我们_应用阈值_来确定哪些像素的强度高于某个特定值,哪些低于该值。
image_binary = image_nuclei > 60
imshow(image_binary)
<matplotlib.image.AxesImage at 0x1dcf6bb09a0>
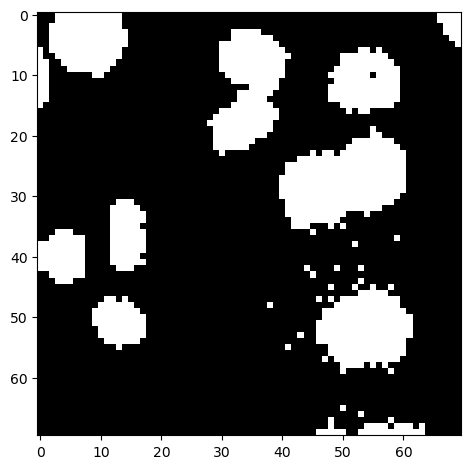
改善二值化结果#
有时,二值化结果可能看起来像素化。通过在阈值化图像之前应用滤波器可以改善这一点。
image_denoised = gaussian(image_nuclei, sigma=1, preserve_range=True)
imshow(image_denoised, cmap='Greys_r')
C:\Users\haase\mambaforge\envs\bio39\lib\site-packages\skimage\io\_plugins\matplotlib_plugin.py:150: UserWarning: Float image out of standard range; displaying image with stretched contrast.
lo, hi, cmap = _get_display_range(image)
<matplotlib.image.AxesImage at 0x1dcf6c48640>

image_binary2 = image_denoised > 60
imshow(image_binary2)
<matplotlib.image.AxesImage at 0x1dcf7d61040>