预告片:使用Python进行生物图像分析#
在接下来的章节中,我们将深入研究使用Python进行图像分析、机器学习和生物统计。这第一个notebook作为我们将要做的事情的预告片。
Python notebook通常以导入notebook将使用的Python库开始。读者可以首先检查是否所有这些库都已安装,然后再浏览整个notebook。
import numpy as np
from skimage.io import imread, imshow
import pyclesperanto_prototype as cle
from skimage import measure
import pandas as pd
import seaborn
import apoc
import stackview
处理图像数据#
我们首先加载感兴趣的图像数据。在这个例子中,我们加载了一张显示斑马鱼眼睛的图像,感谢Mauricio Rocha Martins, Norden实验室, MPI CBG德累斯顿。
# open an image file
multichannel_image = imread("../../data/zfish_eye.tif")
# extract a channel
single_channel_image = multichannel_image[:,:,0]
cropped_image = single_channel_image[200:600, 500:900]
stackview.insight(cropped_image)
|
|
|
图像滤波#
处理荧光显微镜图像时,一个常见的步骤是减去背景强度,例如由焦外光产生的背景强度。这可以进一步改善工作流程中的图像分割结果。
# subtract background using a top-hat filter
background_subtracted_image = cle.top_hat_box(cropped_image, radius_x=20, radius_y=20)
stackview.insight(background_subtracted_image)
|
|
|
图像分割#
对于给定图像中的细胞核分割,存在大量算法。这里我们使用一种名为Voronoi-Otsu标记的经典方法,虽然它并不完美。
label_image = np.asarray(cle.voronoi_otsu_labeling(background_subtracted_image, spot_sigma=4))
# show result
stackview.insight(label_image)
|
|
|
测量和特征提取#
图像分割后,我们可以测量单个对象的属性。这些属性通常是描述性统计参数,称为特征。当我们得出面积或平均强度等测量结果时,我们就提取了这两个特征。
statistics = measure.regionprops_table(label_image,
intensity_image=cropped_image,
properties=('area', 'mean_intensity', 'major_axis_length', 'minor_axis_length'))
处理表格#
上面创建的statistics对象包含一个Python数据结构,即测量向量的字典,这不是最直观的查看方式。因此,我们将其转换为表格。数据科学家经常将这些表格称为DataFrame,它们在pandas库中可用。
dataframe = pd.DataFrame(statistics)
我们可以使用现有的表格列来计算其他测量值,比如aspect_ratio。
dataframe['aspect_ratio'] = dataframe['major_axis_length'] / dataframe['minor_axis_length']
dataframe
| area | mean_intensity | major_axis_length | minor_axis_length | aspect_ratio | |
|---|---|---|---|---|---|
| 0 | 294.0 | 36604.625850 | 25.656180 | 18.800641 | 1.364644 |
| 1 | 91.0 | 37379.769231 | 20.821990 | 6.053507 | 3.439658 |
| 2 | 246.0 | 44895.308943 | 21.830827 | 14.916032 | 1.463581 |
| 3 | 574.0 | 44394.637631 | 37.788705 | 19.624761 | 1.925563 |
| 4 | 518.0 | 45408.903475 | 26.917447 | 24.872908 | 1.082199 |
| ... | ... | ... | ... | ... | ... |
| 108 | 568.0 | 48606.121479 | 37.357606 | 19.808121 | 1.885974 |
| 109 | 175.0 | 25552.074286 | 17.419031 | 13.675910 | 1.273702 |
| 110 | 460.0 | 39031.419565 | 26.138592 | 23.522578 | 1.111213 |
| 111 | 407.0 | 39343.292383 | 28.544027 | 19.563792 | 1.459023 |
| 112 | 31.0 | 29131.322581 | 6.892028 | 5.711085 | 1.206781 |
113 rows × 5 columns
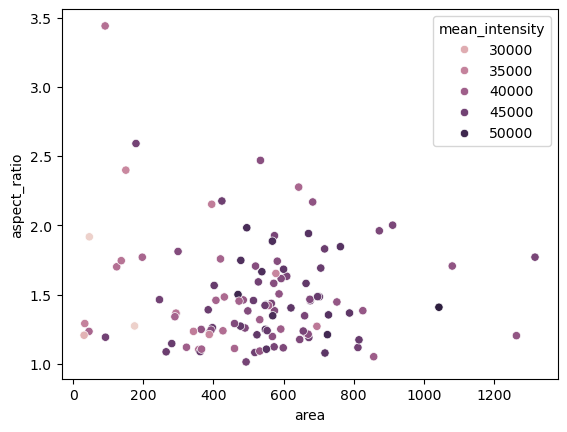
绘图#
可以使用绘图来可视化测量结果。
seaborn.scatterplot(dataframe, x='area', y='aspect_ratio', hue='mean_intensity')
<Axes: xlabel='area', ylabel='aspect_ratio'>
描述性统计#
以此表格为起点,我们可以使用统计数据来概述测量的数据。
mean_area = np.mean(dataframe['area'])
stddev_area = np.std(dataframe['area'])
print("平均细胞核面积为", mean_area, "+-", stddev_area, "像素")
Mean nucleus area is 524.4247787610619 +- 231.74703195433014 pixels
分类#
为了更好地理解组织的内部结构,同时也为了纠正图像处理工作流程中的人为错误,我们可以对细胞进行分类,例如根据它们的大小和形状。
object_classifier = apoc.ObjectClassifier('../../data/blobs_classifier.cl')
classification_image = object_classifier.predict(label_image, cropped_image)
stackview.imshow(classification_image)

