卷积#
当我们对图像应用所谓的_线性_滤波器时,我们将每个新像素计算为其邻居的加权和。这个过程被称为卷积,定义权重的矩阵被称为_卷积核_。在显微镜领域,我们经常谈论显微镜的点扩散函数(PSF)。从技术上讲,这个PSF描述了图像在我们保存到磁盘之前如何被显微镜卷积。
import numpy as np
import pyclesperanto_prototype as cle
from skimage.io import imread
from pyclesperanto_prototype import imshow
from skimage import filters
from skimage.morphology import ball
from scipy.ndimage import convolve
import matplotlib.pyplot as plt
cle.select_device('RTX')
<gfx90c on Platform: AMD Accelerated Parallel Processing (2 refs)>
为了演示卷积的原理,我们首先定义一个相当简单的示例图像。
image = np.asarray([
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 1, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 2, 0, 0],
[0, 0, 0, 0, 0, 0, 1, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0]
]).astype(float)
imshow(image)

接下来,我们定义一个简单的卷积核,它由一个小图像表示。
kernel = np.asarray([
[0, 1, 0],
[1, 1, 1],
[0, 1, 0],
])
接下来,我们使用scipy.ndimage.convolve对图像进行卷积。当我们打印结果时,我们可以看到原始图像中的1是如何扩散的,因为对于每个直接相邻的像素,核会对邻居强度进行求和。如果原始图像中有多个强度 > 0 的像素,结果图像将在它们的邻域计算总和。你可以称这个核为局部求和核。
convolved = convolve(image, kernel)
imshow(convolved, colorbar=True)
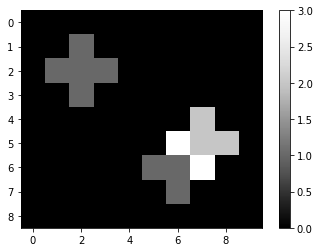
convolved
array([[0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[0., 1., 1., 1., 0., 0., 0., 0., 0., 0.],
[0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[0., 0., 0., 0., 0., 0., 0., 2., 0., 0.],
[0., 0., 0., 0., 0., 0., 3., 2., 2., 0.],
[0., 0., 0., 0., 0., 1., 1., 3., 0., 0.],
[0., 0., 0., 0., 0., 0., 1., 0., 0., 0.],
[0., 0., 0., 0., 0., 0., 0., 0., 0., 0.]])
其他核#
根据用于卷积的核的不同,图像可能看起来非常不同。例如,_均值_核在局部计算平均像素强度:
mean_kernel = np.asarray([
[0, 0.2, 0],
[0.2, 0.2, 0.2],
[0, 0.2, 0],
])
mean_convolved = convolve(image, mean_kernel)
imshow(mean_convolved, colorbar=True)
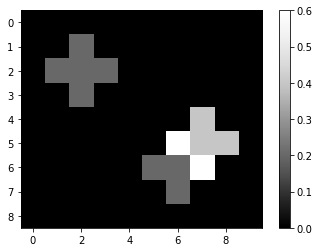
mean_convolved
array([[0. , 0. , 0. , 0. , 0. , 0. , 0. , 0. , 0. , 0. ],
[0. , 0. , 0.2, 0. , 0. , 0. , 0. , 0. , 0. , 0. ],
[0. , 0.2, 0.2, 0.2, 0. , 0. , 0. , 0. , 0. , 0. ],
[0. , 0. , 0.2, 0. , 0. , 0. , 0. , 0. , 0. , 0. ],
[0. , 0. , 0. , 0. , 0. , 0. , 0. , 0.4, 0. , 0. ],
[0. , 0. , 0. , 0. , 0. , 0. , 0.6, 0.4, 0.4, 0. ],
[0. , 0. , 0. , 0. , 0. , 0.2, 0.2, 0.6, 0. , 0. ],
[0. , 0. , 0. , 0. , 0. , 0. , 0.2, 0. , 0. , 0. ],
[0. , 0. , 0. , 0. , 0. , 0. , 0. , 0. , 0. , 0. ]])
以下核是拉普拉斯算子的一种简单形式。
laplace_operator = np.asarray([
[0, 1, 0],
[1, -4, 1],
[0, 1, 0],
])
laplacian = convolve(image, laplace_operator)
imshow(laplacian, colorbar=True)

laplacian
array([[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 1., -4., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 2., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 3., -8., 2., 0.],
[ 0., 0., 0., 0., 0., 1., -4., 3., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.]])
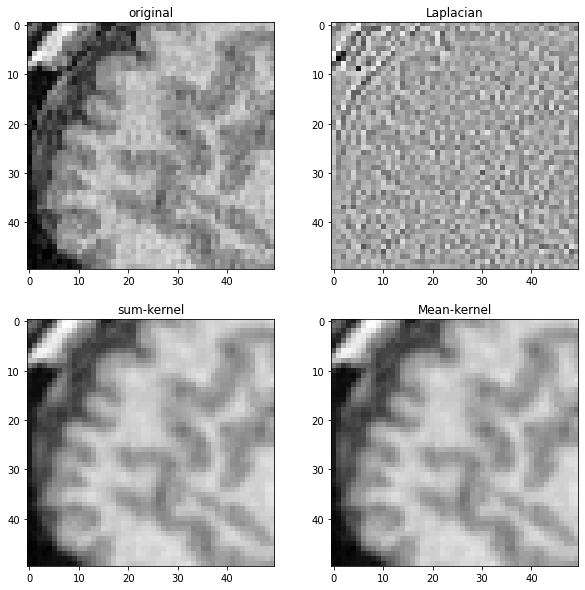
为了演示这些不同的核的作用,我们将它们应用于之前显示的MRI图像。
# 打开数据集并提取单个平面
noisy_mri = imread('../../data/Haase_MRT_tfl3d1.tif')[90].astype(float)
# 通过裁剪一部分来放大
noisy_mri_zoom = noisy_mri[50:100, 50:100]
convolved_mri = convolve(noisy_mri_zoom, kernel)
mean_mri = convolve(noisy_mri_zoom, mean_kernel)
laplacian_mri = convolve(noisy_mri_zoom, laplace_operator)
fig, axes = plt.subplots(2, 2, figsize=(10,10))
imshow(noisy_mri_zoom, plot=axes[0,0])
axes[0,0].set_title("原始")
imshow(laplacian_mri, plot=axes[0,1])
axes[0,1].set_title("拉普拉斯")
imshow(convolved_mri, plot=axes[1,0])
axes[1,0].set_title("求和核")
imshow(mean_mri, plot=axes[1,1])
axes[1,1].set_title("均值核")
Text(0.5, 1.0, 'Mean-kernel')