三维邻居网格#
我们在本章中处理的邻居网格通常也适用于三维。只是在可视化方面更具挑战性。
from skimage.io import imread
import pyclesperanto_prototype as cle
import matplotlib.pyplot as plt
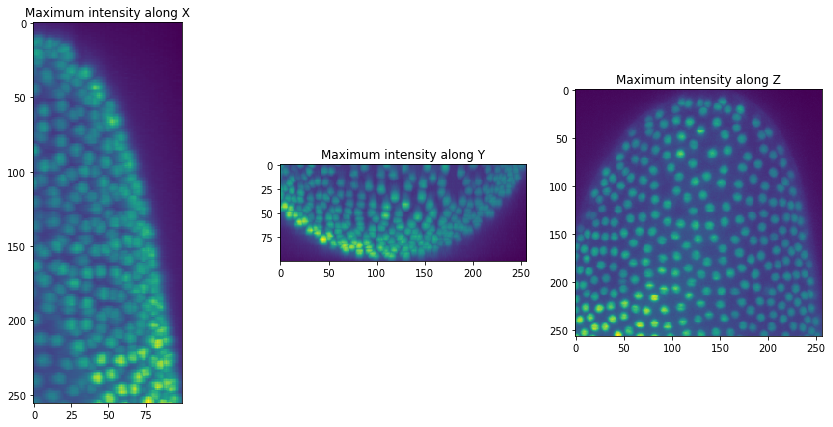
我们在这里使用的图像数据显示了一个发育中的谷斑皮蠹(Tribolium castaneum)胚胎的局部,这是由德累斯顿MPI-CBG / CSBD的Myers实验室的Daniela Vorkel使用光片显微镜拍摄的。
raw_image = imread("../../data/Lund_000500_resampled-cropped.tif")
raw_image.shape
(100, 256, 256)
为了便于使用,我们编写了一个简短的函数,用于从不同角度以三个最大投影来可视化我们的图像堆栈。
def orthogonal_show(image, labels=False):
fig, axs = plt.subplots(1, 3, figsize=(15, 7))
cle.imshow(cle.maximum_x_projection(image), plot=axs[0], labels=labels)
cle.imshow(cle.maximum_y_projection(image), plot=axs[1], labels=labels)
cle.imshow(cle.maximum_z_projection(image), plot=axs[2], labels=labels)
axs[0].set_title("沿X轴的最大强度")
axs[1].set_title("沿Y轴的最大强度")
axs[2].set_title("沿Z轴的最大强度")
orthogonal_show(raw_image)
现在我们可以对数据集中的细胞核进行分割。
background_subtracted = cle.top_hat_box(raw_image, radius_x=5, radius_y=5, radius_z=5)
nuclei = cle.voronoi_otsu_labeling(background_subtracted)
orthogonal_show(nuclei, labels=True)
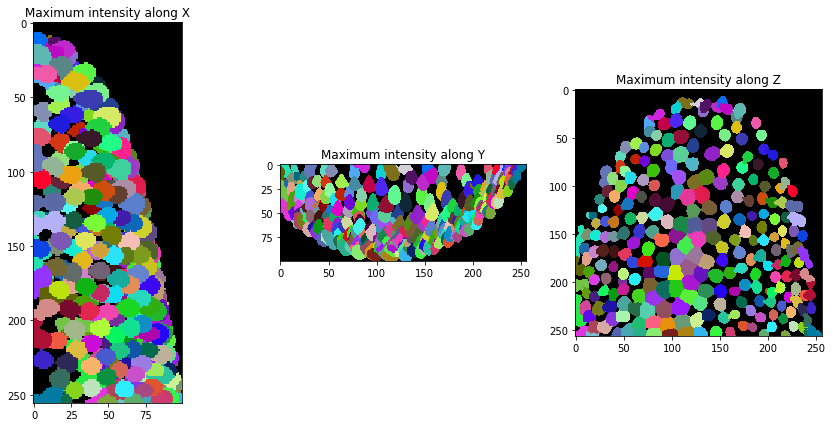
分割后,我们稍微扩展一下标签,使它们相互接触。
expanded_nuclei = cle.dilate_labels(nuclei, radius=4)
orthogonal_show(expanded_nuclei, labels=True)
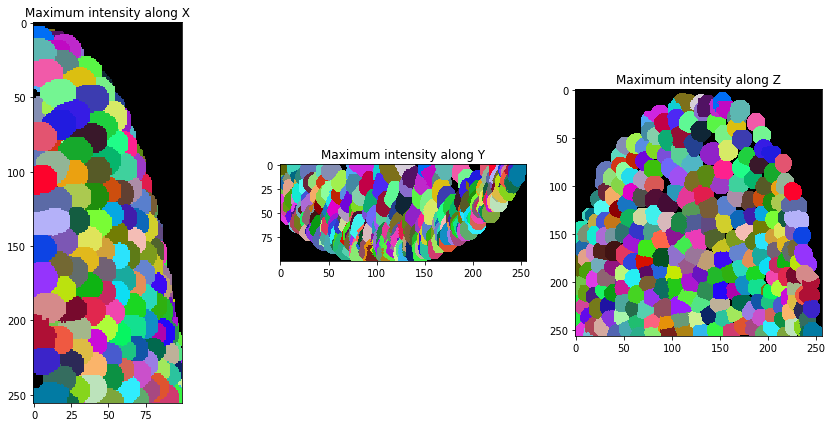
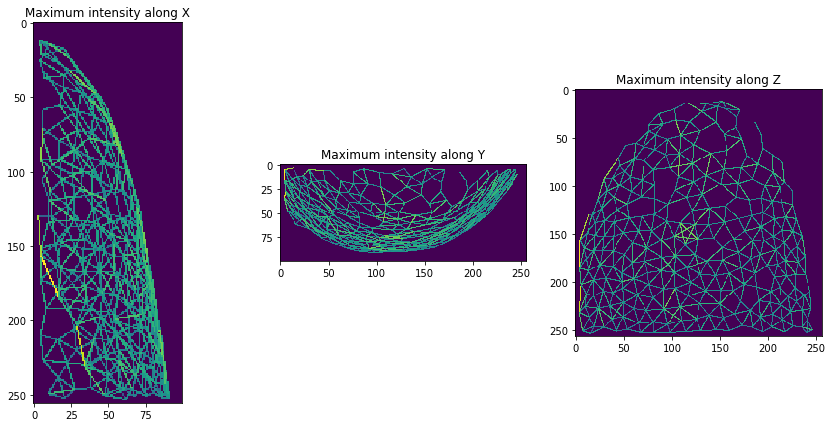
然后,我们可以可视化接触邻居之间的(质心)距离网格。
mesh = cle.draw_distance_mesh_between_touching_labels(expanded_nuclei)
orthogonal_show(mesh)