图像反卷积简介#
在显微镜成像中,由于光学系统(即显微镜)的特性,图像通常会发生失真。发射光子的生物结构在场景图像中不会100%地反映现实。图像会变得模糊,与光学设置的点扩散函数(PSF)进行卷积。如果我们知道PSF,从技术上讲,可以在某种程度上_撤销_这种卷积。
import numpy as np
from skimage.io import imread, imsave
from skimage.data import cells3d
from skimage.filters import gaussian
from scipy.ndimage import convolve
from pyclesperanto_prototype import imshow
import pyclesperanto_prototype as cle
from napari_simpleitk_image_processing import richardson_lucy_deconvolution
import matplotlib.pyplot as plt
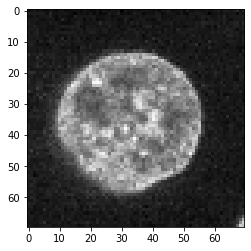
为了演示卷积和反卷积,我们暂时假设这张显示细胞核的图像反映了生物样本中的实际情况。
image = cells3d()[30,1,120:190,80:150]
imshow(image)
此外,我们构造一个人工PSF图像。我们从一个完美的PSF图像开始,除了一个像素外,所有像素都是黑色(0),通常但不必要在中心。
perfect_psf = np.zeros( (25,25) )
perfect_psf[12, 12] = 1
imshow(perfect_psf, colorbar=True)

注意,在上面的例子中,图像的总强度为1。接下来显示的模糊图像的总强度也将接近1。这在后面进行反卷积时至关重要,因为这使得图像(或任何成像的生物结构)的总强度通过反卷积不会改变。
从这个完美的PSF,我们通过高斯模糊得到一个更加实际的PSF示例。
psf = gaussian(perfect_psf, sigma=2)
imshow(psf, colorbar=True)
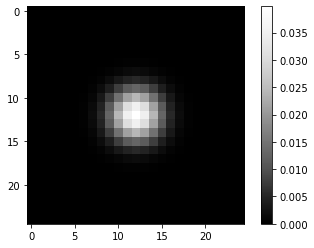
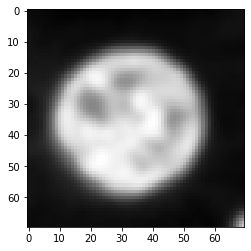
卷积#
正如在图像滤波一节中介绍的那样,卷积是将给定输入图像的每个像素值与根据PSF加权的邻近像素结合的过程。
convolved = convolve(image, psf)
imshow(convolved)
反卷积#
从卷积图像恢复原始图像的一种常见算法是Richardson-Lucy反卷积,它在SimpleITK的RichardsonLucyDeconvolutionImageFilter中有实现。这里我们使用[napari-simpleitk-image-processing]中的一个便捷层。
number_of_iterations = 10
deconvolved = richardson_lucy_deconvolution(convolved, psf, number_of_iterations)
imshow(deconvolved)
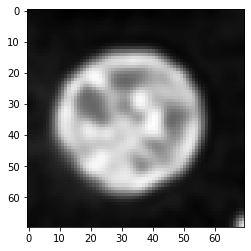
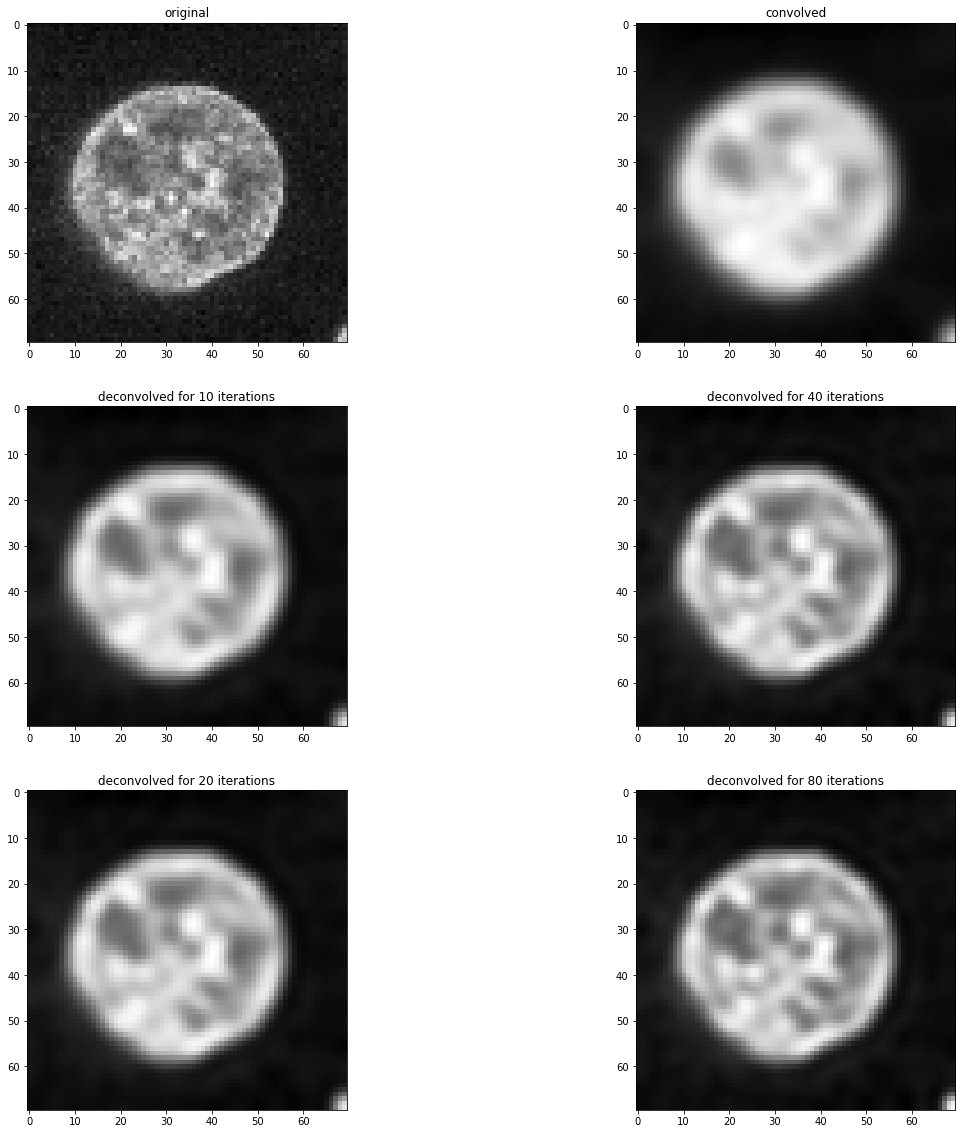
如你所见,这个反卷积后的图像与上面显示的原始图像不相等。反卷积不是魔法。它是一个图像处理滤波器,所有滤波器都有局限性。此外,对于这个反卷积算法,结果取决于迭代次数。我们可以通过测试不同的值来经验性地找到这个参数的好的数值。
fig, axs = plt.subplots(3, 2, figsize=(20,20))
imshow(image, plot=axs[0, 0])
axs[0,0].set_title("原始图像")
imshow(convolved, plot=axs[0, 1])
axs[0,1].set_title("卷积后")
for i, number_of_iterations in enumerate([10, 20, 40, 80]):
deconvolved = richardson_lucy_deconvolution(convolved, psf, number_of_iterations)
axis = axs[(i) % 2 + 1, int((i) / 2)]
imshow(deconvolved, plot=axis)
axis.set_title("反卷积 " + str(number_of_iterations) + " 次迭代")
练习#
编写一个for循环,使用不同的number_of_iterations对convolved图像进行反卷积,并确定原始图像与反卷积图像之间的均方误差。
提示:你可以使用sklearn.metrics.mean_squared_error。
