Optimierungsgrundlagen#
In diesem Notebook zeigen wir, wie man einen Workflow zur Bildsegmentierung einrichtet und dessen Parameter mit einer gegebenen spärlichen Annotation optimiert.
Siehe auch:
from skimage.io import imread
from scipy.optimize import minimize
import numpy as np
import pyclesperanto_prototype as cle
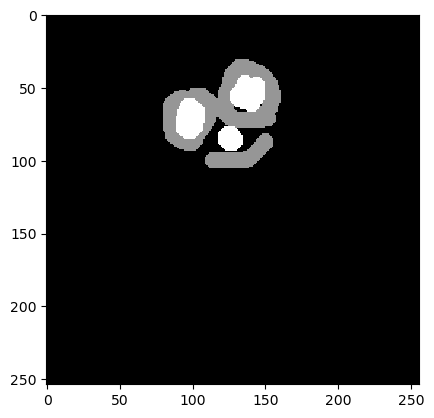
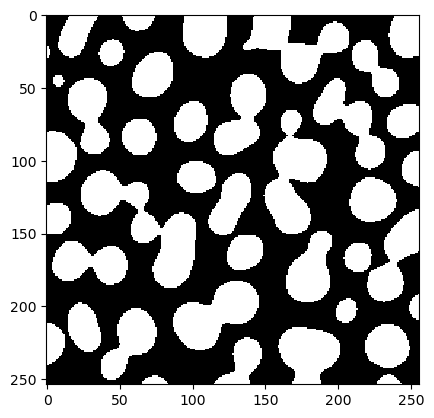
Wir beginnen mit dem Laden eines Beispielbildes und einer manuellen Annotation. Es müssen nicht alle Objekte annotiert sein (spärliche Annotation).
blobs = imread('../../data/blobs.tif')
cle.imshow(blobs)

annotation = imread('../../data/blobs_annotated.tif')
cle.imshow(annotation)
Als Nächstes definieren wir einen Bildverarbeitungs-Workflow, der zu einem Binärbild führt.
def workflow(image, sigma, threshold):
blurred = cle.gaussian_blur(image, sigma_x=sigma, sigma_y=sigma)
binary = cle.greater_constant(blurred, constant=threshold)
return binary
Wir testen diesen Workflow auch mit einigen zufälligen sigma und threshold Werten.
test = workflow(blobs, 5, 100)
cle.imshow(test)
Unsere Fitnessfunktion nimmt zwei Parameter: Ein gegebenes Segmentierungsergebnis (test) und eine Referenzannotation. Sie bestimmt dann, wie gut die Segmentierung ist, z.B. unter Verwendung des Jaccard-Index.
binary_and = cle.binary_and
def fitness(test, reference):
"""
Bestimmt, wie korrekt eine gegebene Testsegmentierung ist.
Als Metrik verwenden wir den Jaccard-Index.
Annahme: test ist ein Binärbild (0=Falsch und 1=Wahr) und
reference ist ein Bild mit 0=unbekannt, 1=Falsch, 2=Wahr.
"""
negative_reference = reference == 1
positive_reference = reference == 2
negative_test = test == 0
positive_test = test == 1
# true positive: test = 1, ref = 2
tp = binary_and(positive_reference, positive_test).sum()
# true negative:
tn = binary_and(negative_reference, negative_test).sum()
# false positive
fp = binary_and(negative_reference, positive_test).sum()
# false negative
fn = binary_and(positive_reference, negative_test).sum()
# return Jaccard Index
return tp / (tp + fn + fp)
fitness(test, annotation)
0.74251497
Wir sollten diese Funktion auch für eine Reihe von Parametern testen.
sigma = 5
for threshold in range(70, 180, 10):
test = workflow(blobs, sigma, threshold)
print(threshold, fitness(test, annotation))
70 0.49048626
80 0.5843038
90 0.67019403
100 0.74251497
110 0.8183873
120 0.8378158
130 0.79089373
140 0.7024014
150 0.60603446
160 0.49827588
170 0.3974138
Als Nächstes definieren wir eine funktion, die nur numerische Parameter enthält, die optimiert werden sollen.
def fun(x):
# aktuelle Parametereinstellung anwenden
test = workflow(blobs, x[0], x[1])
# da wir minimieren, multiplizieren wir die Fitness mit -1
return -fitness(test, annotation)
Bevor wir mit der Optimierung beginnen, besteht der letzte Schritt darin, den Startpunkt x0 für die Optimierung und das Abbruchkriterium atol, den absoluten Toleranzwert, zu konfigurieren.
# Startpunkt im Parameterraum
x0 = np.array([5, 100])
# Optimierung durchführen
result = minimize(fun, x0, method='nelder-mead', options={'xatol': 1e-3})
result
final_simplex: (array([[ 3.89501953, 121.94091797],
[ 3.89498663, 121.9409585 ],
[ 3.89500463, 121.9403702 ]]), array([-0.85761315, -0.85761315, -0.85761315]))
fun: -0.8576131463050842
message: 'Optimization terminated successfully.'
nfev: 65
nit: 22
status: 0
success: True
x: array([ 3.89501953, 121.94091797])
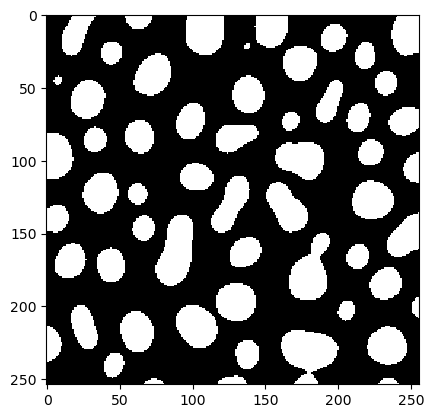
Aus diesem result-Objekt können wir den Parametersatz auslesen, der als optimal ermittelt wurde, und ein Binärbild erzeugen.
x = result['x']
best_binary = workflow(blobs, x[0], x[1])
cle.imshow(best_binary)
Eine Anmerkung zur Konvergenz#
Optimierungsalgorithmen finden möglicherweise nicht immer das globale Optimum. Der Erfolg hängt vom Startpunkt der Optimierung, von der Form des Parameterraums und dem gewählten Algorithmus ab. Im folgenden Beispiel zeigen wir, wie eine fehlgeschlagene Optimierung aussehen kann, wenn der Startpunkt schlecht gewählt wurde.
# Startpunkt im Parameterraum
x0 = np.array([0, 60])
# Optimierung durchführen
result = minimize(fun, x0, method='nelder-mead', options={'xatol': 1e-3})
result
final_simplex: (array([[0.00000000e+00, 6.00000000e+01],
[6.10351563e-08, 6.00000000e+01],
[0.00000000e+00, 6.00007324e+01]]), array([-0.63195992, -0.63195992, -0.63195992]))
fun: -0.6319599151611328
message: 'Optimization terminated successfully.'
nfev: 51
nit: 13
status: 0
success: True
x: array([ 0., 60.])
Fehlerbehebung: Erkundung des Parameterraums#
In diesem Fall unterscheidet sich der resultierende Parametersatz nicht vom Startpunkt. Falls sich die Fitness um den Startpunkt herum nicht ändert, weiß der Optimierungsalgorithmus nicht, wie er das Ergebnis verbessern soll. Die Visualisierung der Werte um den Startpunkt herum kann helfen.
sigma = 0
for threshold in range(57, 63):
test = workflow(blobs, sigma, threshold)
print(threshold, fitness(test, annotation))
57 0.6319599
58 0.6319599
59 0.6319599
60 0.6319599
61 0.6319599
62 0.6319599
threshold = 60
for sigma in np.arange(0, 0.5, 0.1):
test = workflow(blobs, sigma, threshold)
print(sigma, fitness(test, annotation))
0.0 0.6319599
0.1 0.6319599
0.2 0.6319599
0.30000000000000004 0.6319599
0.4 0.6319599
Daher ist es sinnvoll, vor der automatischen Optimierung eine manuelle Erkundung des Parameterraums durchzuführen.
