Schwellenwertverfahren#
Schwellenwertverfahren ist eine Technik der Bildsegmentierung. Es trennt ein gegebenes Einzelkanal-Bild (oder einen Stapel) in zwei Bereiche: Pixel mit Intensität unterhalb eines gegebenen Schwellenwerts, auch “Hintergrund” genannt, und Pixel mit Intensität oberhalb eines gegebenen Schwellenwerts, “Vordergrund”. Typischerweise resultieren diese Algorithmen in Binärbildern, bei denen die Hintergrundintensität 0 und die Vordergrundintensität 1 ist. Bei der Anwendung solcher Algorithmen in ImageJ haben Vordergrundpixel den Wert 255. In scikit-image sind Hintergrundpixel False und Vordergrundpixel True.
Siehe auch
from skimage.io import imread
from pyclesperanto_prototype import imshow
import pyclesperanto_prototype as cle
from skimage import filters
from skimage.filters import try_all_threshold
from matplotlib import pyplot as plt
import napari_simpleitk_image_processing as nsitk
image = imread("../../data/blobs.tif")
imshow(image)
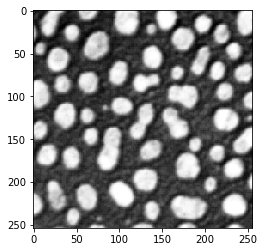
Bildsegmentierung durch Schwellenwertverfahren#
Die threshold_otsu Operation, auch bekannt als Otsu’s Methode (Otsu et al., IEEE Transactions on Systems, Man, and Cybernetics, Vol. 9 (1), 1979), liefert eine Zahl - den anzuwendenden Schwellenwert.
threshold = filters.threshold_otsu(image)
Bei der Verwendung von Methoden wie Schwellenwertverfahren in Notebooks wird empfohlen, das Ergebnis auszugeben, um zu sehen, was es tatsächlich zurückgibt. Hier verwenden wir die Methode von scikit-image, die den anzuwendenden Schwellenwert zurückgibt. Das Ausdrucken dieses Schwellenwerts kann später bei der Reproduktion des Workflows hilfreich sein, auch wenn andere den gleichen Schwellenwert auf den Datensatz in anderer Software anwenden möchten.
threshold
120
Mit numpy-Arrays können wir den Schwellenwert anwenden, indem wir den >=-Operator verwenden. Das Ergebnis wird ein Binärbild sein.
binary_image = image >= threshold
imshow(binary_image)

Wir können auch bestimmen, in welchem Typ das Binärbild verarbeitet wird, indem wir Minimum und Maximum des Bildes ausgeben:
binary_image.max()
True
binary_image.min()
False
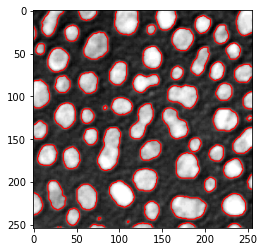
Wie bereits gezeigt, erlaubt uns matplotlib, eine Kontur auf ein mit imshow visualisiertes Bild zu zeichnen, indem wir den contour-Befehl verwenden.
# Erstelle einen neuen Plot
fig, axes = plt.subplots(1,1)
# Füge zwei Bilder hinzu
axes.imshow(image, cmap=plt.cm.gray)
axes.contour(binary_image, [0.5], linewidths=1.2, colors='r')
<matplotlib.contour.QuadContourSet at 0x2b57076dc70>
Es gibt eine Liste von Schwellenwert-Algorithmen. Es ist möglich, sie alle auf Ihre Daten anzuwenden und die Unterschiede zu sehen:
fig, ax = try_all_threshold(image, figsize=(10, 8), verbose=False)
plt.show()

Schwellenwertverfahren mit pyclesperanto#
Darüber hinaus bieten auch andere Bibliotheken wie pyclesperanto Schwellenwert-Algorithmen an. Die Implementierung hier gibt nicht den Schwellenwert zurück, sondern direkt das Binärbild.
binary_image2 = cle.threshold_otsu(image)
imshow(binary_image2)

Hier können wir auch sehen, dass verschiedene Bibliotheken Binärbilder auf unterschiedliche Weise speichern. pyclesperanto speichert zum Beispiel die positiven Pixel in Binärbildern nicht als True, sondern stattdessen mit einer 1:
binary_image2.max()
1.0
binary_image2.min()
0.0
Schwellenwertverfahren mit SimpleITK#
Auch SimpleITK bietet Schwellenwert-Algorithmen, die in der Liste der Filter zu finden sind. Für die Skript-Bequemlichkeit verwenden wir hier napari-simpleitk-image-processing, ein skriptbares napari-Plugin, das einige SimpleITK-Funktionen auf zugänglichere Weise anbietet. Wir können eine kleine Schleife programmieren, die alle Schwellenwert-Algorithmen in SimpleITK ausprobiert und uns die Ergebnisse zeigt:
threshold_algorithms = [
nsitk.threshold_huang,
nsitk.threshold_intermodes,
nsitk.threshold_isodata,
nsitk.threshold_kittler_illingworth,
nsitk.threshold_li,
nsitk.threshold_maximum_entropy,
nsitk.threshold_moments,
nsitk.threshold_otsu,
nsitk.threshold_renyi_entropy,
nsitk.threshold_shanbhag,
nsitk.threshold_triangle,
nsitk.threshold_yen
]
for algorithm in threshold_algorithms:
# Zeige den Namen des Algorithmus über dem Bild
print(algorithm.__name__)
# Binarisiere das Bild mit dem gegebenen Algorithmus
binary_image = algorithm(image)
# Zeige das Segmentierungsergebnis
imshow(binary_image)
threshold_huang

threshold_intermodes

threshold_isodata

threshold_kittler_illingworth

threshold_li

threshold_maximum_entropy

threshold_moments

threshold_otsu

threshold_renyi_entropy

threshold_shanbhag

threshold_triangle

threshold_yen

Übung#
Segmentieren Sie blobs.tif mit dem Yen-Algorithmus. Verwenden Sie matplotlib, um eine grüne Kontur der segmentierten Objekte um die Regionen auf dem Originalbild zu zeichnen.
Segmentieren Sie das Bild mit einem berechneten Schwellenwert gemäß dieser Gleichung:
threshold = mean + 2 * standard_deviation
Visualisieren Sie das resultierende Segmentierungsergebnis mit einer roten Kontur auf dem Originalbild und der grünen Kontur von oben.
Alternativ können Sie beide Segmentierungsergebnisse in napari einfügen und dort visuell vergleichen.
