Öffnen von LIF-Dateien#
Bei der Arbeit mit mikroskopischen Bilddaten sind viele Dateiformate im Umlauf, wie zum Beispiel das Leica Image Format (LIF). In diesem Notebook werden wir eine .lif-Datei mit Hilfe der readlif Bibliothek öffnen.
Hinweis: Es wird empfohlen, AICSImageIO zum Lesen von LIF-Bildern zu verwenden, wie in diesem Notebook gezeigt.
Die readlif Bibliothek kann wie folgt über das Terminal installiert werden:
pip install readlif
Nach der Installation kann sie importiert werden.
from readlif.reader import LifFile
import os
import requests
from skimage.io import imshow
import numpy as np
Als Beispieldatensatz verwenden wir ein Bild, das von Gregory Marquart und Harold Burgess unter der CC-BY 4.0 Lizenz geteilt wurde. Wir müssen es zuerst herunterladen.
filename = "../../data/y293-Gal4_vmat-GFP-f01.lif"
url = 'https://zenodo.org/record/3382102/files/y293-Gal4_vmat-GFP-f01.lif?download=1'
if not os.path.isfile(filename):
# die Datei nur herunterladen, wenn wir sie noch nicht haben
response = requests.get(url)
open(filename, "wb").write(response.content)
An diesem Punkt sollte die Datei auf unserem Computer sein und kann wie folgt geöffnet werden.
file = LifFile(filename)
file
'LifFile object with 1 image'
lif_image = file.get_image(0)
lif_image
'LifImage object with dimensions: Dims(x=616, y=500, z=86, t=1, m=1)'
Aus dem LifImage können wir einzelne Frames als PIL Bilder erhalten.
pil_image = lif_image.get_frame(z=0)
type(pil_image)
PIL.Image.Image
Schließlich können diese 2D PIL-Bilder in numpy-Arrays umgewandelt werden. Das ermöglicht es uns letztendlich, einen Blick auf das Bild zu werfen.
np_image = np.array(pil_image)
np_image.shape
(500, 616)
imshow(np_image)
/opt/miniconda3/envs/devbio-napari-env/lib/python3.9/site-packages/skimage/io/_plugins/matplotlib_plugin.py:149: UserWarning: Low image data range; displaying image with stretched contrast.
lo, hi, cmap = _get_display_range(image)
<matplotlib.image.AxesImage at 0x1378ab970>

Um auf alle Pixel in unserem 3D-Bild zuzugreifen, sollten wir zunächst einen Blick in die Metadaten der Datei werfen.
lif_image.info
{'dims': Dims(x=616, y=500, z=86, t=1, m=1),
'display_dims': (1, 2),
'dims_n': {1: 616, 2: 500, 3: 86},
'scale_n': {1: 2.1354804344851965,
2: 2.135480168493237,
3: 0.9929687300128537},
'path': 'Experiment_002/',
'name': 'Series011',
'channels': 2,
'scale': (2.1354804344851965, 2.135480168493237, 0.9929687300128537, None),
'bit_depth': (12, 12),
'mosaic_position': [],
'channel_as_second_dim': False,
'settings': {}}
Zum Beispiel könnte es später nützlich sein, die Voxelgröße in z/y/x-Reihenfolge zu kennen.
voxel_size = lif_image.info['scale'][2::-1]
voxel_size
(0.9929687300128537, 2.135480168493237, 2.1354804344851965)
Wir können auch auslesen, wie viele Schichten der 3D-Stapel hat.
num_slices = lif_image.info['dims'].z
num_slices
86
Diese Information ermöglicht es uns, eine Hilfsfunktion zu schreiben, die das LIF-Bild in einen 3D-numpy-Bildstapel umwandelt.
def lif_to_numpy_stack(lif_image):
num_slices = lif_image.info['dims'].z
return np.asarray([np.array(lif_image.get_frame(z=z)) for z in range(num_slices)])
image_stack = lif_to_numpy_stack(lif_image)
image_stack.shape
(86, 500, 616)
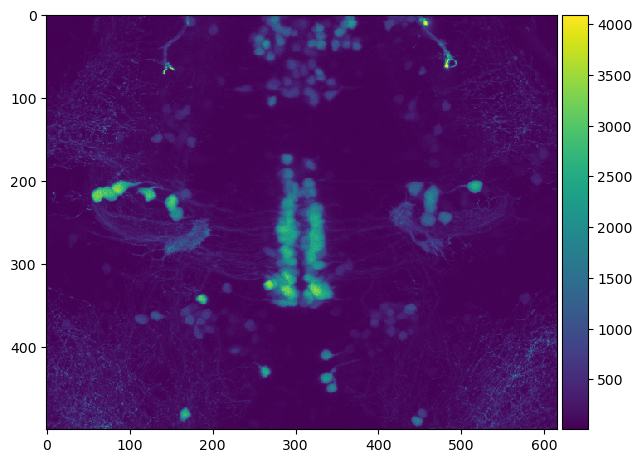
Dieser Bildstapel kann dann beispielsweise verwendet werden, um eine Maximumintensitätsprojektion entlang Z zu visualisieren.
imshow(np.max(image_stack, axis=0))
<matplotlib.image.AxesImage at 0x137a1c610>