Formdeskriptoren basierend auf Nachbarschaftsgraphen#
Dieses Notebook zeigt, wie man Formdeskriptoren von Zellen bestimmen kann, wenn sie nicht genau segmentiert werden können, aber ihre Zentren erkannt werden können.
from skimage.measure import regionprops
import numpy as np
import pyclesperanto_prototype as cle
cle.get_device()
<gfx90c on Platform: AMD Accelerated Parallel Processing (2 refs)>
Wir erzeugen ein Labelbild von Zellen mit gegebenen Größen in x und y und einem Größenverhältnis von 1:1,5.
Nehmen wir an, dies ist das Ergebnis eines Zellsegmentierungsalgorithmus.
cell_size_x = 10
cell_size_y = 15
# generate and show tissue
tissue_labels = cle.artificial_tissue_2d(width=100, height=100, delta_x=cell_size_x, delta_y=cell_size_y, random_sigma_x=1, random_sigma_y=1)
cle.imshow(tissue_labels, labels=True)

Klassische Formdeskriptoren: kleine und große Achse#
Wir können die kleine und große Achse dieser Zellen mit scikit-image messen
label_image = cle.pull_zyx(tissue_labels).astype(int)
stats = regionprops(label_image)
avg_minor_axis_length = np.mean([s.minor_axis_length for s in stats])
print("Durchschnittliche Länge der kleinen Achse", avg_minor_axis_length)
avg_major_axis_length = np.mean([s.major_axis_length for s in stats])
print("Durchschnittliche Länge der großen Achse", avg_major_axis_length)
Average minor axis length 11.094738115993875
Average major axis length 16.966309163176998
Wir erzeugen nun ein Bild, bei dem die Zellen möglicherweise schwer zu segmentieren sind, z.B. ein Membranbild.
cell_borders = cle.detect_label_edges(label_image)
convolved_membranes = cle.gaussian_blur(cell_borders, sigma_x=1, sigma_y=2)
noise_level = 3
noise = (np.random.random(convolved_membranes.shape) - 0.5) * cle.mean_of_all_pixels(convolved_membranes) * noise_level
noise = noise.astype(np.float32)
artifical_membrane_image = (convolved_membranes + noise)
cle.imshow(artifical_membrane_image)

Formdeskriptoren basierend auf Nachbarschaftsnetzen#
In manchen Fällen können wir die Zellen nicht richtig segmentieren, wir können nur eine Punkterkennung durchführen und die Zentren der Zellen visualisieren.
blurred = cle.gaussian_blur(artifical_membrane_image, sigma_x=2, sigma_y=2)
local_minima = cle.detect_minima_box(blurred)
spot_image = cle.label_spots(local_minima)
# we extend the spots a little bit for visualization purposes
spot_image = cle.maximum_sphere(spot_image, radius_x=1, radius_y=1)
cle.imshow(spot_image, labels=True)

Aus einem solchen Bild von markierten Punkten können wir ein Voronoi-Diagramm erstellen, in dem wir analysieren können, welche Zellen (erweiterte Punkte) nahe beieinander liegen.
Das Ergebnis ist eine Annäherung an die Zellsegmentierung.
voronoi_diagram_all = cle.extend_labeling_via_voronoi(spot_image)
# exclude labels on image edges
voronoi_diagram = cle.exclude_labels_on_edges(voronoi_diagram_all)
cle.imshow(voronoi_diagram, labels=True)

Aus einem solchen Paar von Punktbild und Voronoi-Diagramm können wir zwei Matrizen ableiten: eine Berührungsmatrix (auch bekannt als Adjazenzgraphmatrix) und eine Distanzmatrix.
touch_matrix = cle.generate_touch_matrix(voronoi_diagram)
# igonore touching the background
cle.set_column(touch_matrix, 0, 0)
centroids = cle.centroids_of_labels(voronoi_diagram)
distance_matrix = cle.generate_distance_matrix(centroids, centroids)
cle.imshow(touch_matrix)
cle.imshow(distance_matrix)


Aus diesen beiden Matrizen können wir den minimalen und maximalen Abstand zwischen den Zentroiden von sich berührenden Objekten (Zellen) im Voronoi-Bild bestimmen. Dies sind geschätzte kleine und große Achsen der segmentierten Objekte.
min_distance = cle.minimum_distance_of_touching_neighbors(distance_matrix, touch_matrix)
max_distance = cle.maximum_distance_of_touching_neighbors(distance_matrix, touch_matrix)
print("Minimaler Abstand berührender Nachbarn", cle.mean_of_all_pixels(min_distance))
print("Maximaler Abstand berührender Nachbarn", cle.mean_of_all_pixels(max_distance))
minimum distance of touching neihbors 10.007136855014535
maximum distance of touching neihbors 16.39732325354288
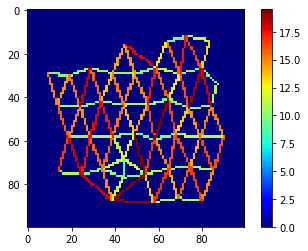
Distanzvisualisierung#
Zum Schluss visualisieren wir die Abstände zwischen Nachbarn in einem farbigen Netz.
mesh = cle.draw_distance_mesh_between_touching_labels(voronoi_diagram)
cle.imshow(mesh, colorbar=True, colormap="jet")