Nachbearbeitung von Binärbildern mit morphologischen Operationen#
Morphologische Operationen transformieren Bilder basierend auf der Form; typischerweise sprechen wir in diesem Kontext von Binärbildern.
import numpy as np
from skimage.io import imread
import matplotlib.pyplot as plt
from skimage import morphology
from skimage import filters
Kerne, Fußabdrücke und strukturelle Elemente#
Wenn wir mit scikit-image arbeiten, haben viele morphologische Filter einen footprint-Parameter. Dieser Fußabdruck ist der Filterkern, und in der Literatur finden Sie auch den Begriff strukturelles Element dafür.
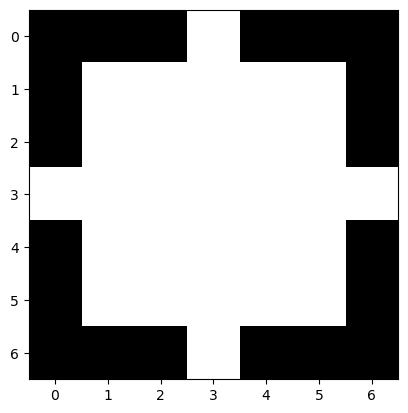
# erstellt eine Scheibe von 1 mit Radius = 3
disk = morphology.disk(3)
disk
array([[0, 0, 0, 1, 0, 0, 0],
[0, 1, 1, 1, 1, 1, 0],
[0, 1, 1, 1, 1, 1, 0],
[1, 1, 1, 1, 1, 1, 1],
[0, 1, 1, 1, 1, 1, 0],
[0, 1, 1, 1, 1, 1, 0],
[0, 0, 0, 1, 0, 0, 0]], dtype=uint8)
plt.imshow(disk, cmap='gray')
<matplotlib.image.AxesImage at 0x225b0c88340>
# erstellt ein Quadrat mit Breite und Höhe = 3
square = morphology.square(3)
square
array([[1, 1, 1],
[1, 1, 1],
[1, 1, 1]], dtype=uint8)
Binäre Morphologie#
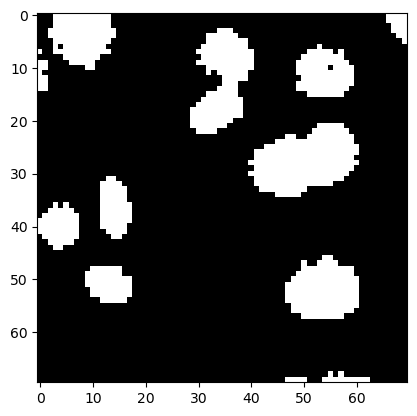
Zur Demonstration der morphologischen Filterung von Binärbildern verwenden wir wieder das kleine Zellkernbild.
image_nuclei = imread('../../data/mitosis_mod.tif').astype(float)
image_binary = image_nuclei > filters.threshold_otsu(image_nuclei)
plt.imshow(image_binary, cmap='gray')
<matplotlib.image.AxesImage at 0x225b0d05490>
Erosion und Dilatation#
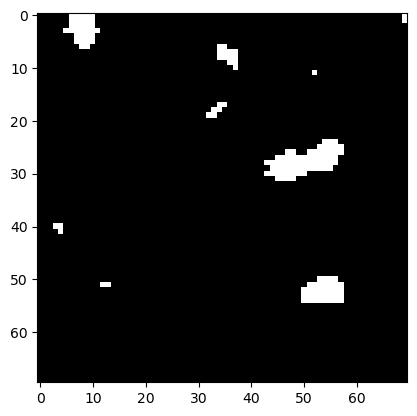
Um weiße Inseln im schwarzen Ozean zu verkleinern, müssen wir ihre Küstenlinien erodieren.
eroded = morphology.binary_erosion(image_binary, disk)
plt.imshow(eroded, cmap='gray')
<matplotlib.image.AxesImage at 0x225b0d80dc0>
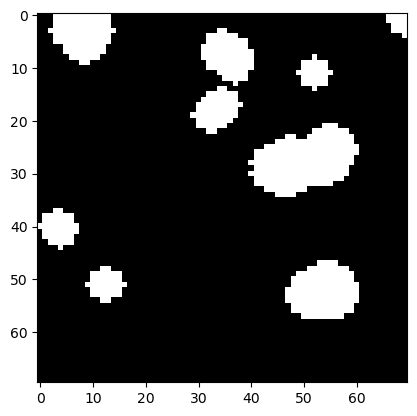
Wenn wir das Bild anschließend dilatieren, erhalten wir weiße Inseln zurück, die glatter aussehen als im ursprünglichen Binärbild.
eroded_dilated = morphology.binary_dilation(eroded, disk)
plt.imshow(eroded_dilated, cmap='gray')
<matplotlib.image.AxesImage at 0x225b107f730>
Das aufeinanderfolgende Aufrufen von Erosion und Dilatation ist so häufig, dass es dafür eine extra Funktion gibt, die genau das macht. Da sich die Lücke zwischen den Inseln öffnet, wird die Operation als Öffnung bezeichnet.
opened = morphology.binary_opening(image_binary, disk)
plt.imshow(opened, cmap='gray')
<matplotlib.image.AxesImage at 0x225b10f9b20>

Übung 1#
Es gibt auch eine Schließungs-Operation. Wenden Sie sie auf das Binärbild an.
Übung 2#
Suchen Sie in der scikit-image Dokumentation nach Minimum- und Maximum-Filtern. Wenden Sie den Minimum-Filter auf das Binärbild und anschließend den Maximum-Filter auf das Ergebnis an. Vergleichen Sie es mit den oben gezeigten Bildern.
