Trailer: Biobildanalyse mit Python#
In den folgenden Kapiteln werden wir uns in die Bildanalyse, maschinelles Lernen und Biostatistik mit Python vertiefen. Dieses erste Notebook dient als Vorschau auf das, was wir machen werden.
Python-Notebooks beginnen typischerweise mit den Importen der Python-Bibliotheken, die das Notebook verwenden wird. Der Leser kann zunächst überprüfen, ob all diese Bibliotheken installiert sind, bevor er das gesamte Notebook durchgeht.
import numpy as np
from skimage.io import imread, imshow
import pyclesperanto_prototype as cle
from skimage import measure
import pandas as pd
import seaborn
import apoc
import stackview
Arbeiten mit Bilddaten#
Wir beginnen mit dem Laden der interessierenden Bilddaten. In diesem Beispiel laden wir ein Bild, das ein Zebrafischauge zeigt, mit freundlicher Genehmigung von Mauricio Rocha Martins, Norden-Labor, MPI CBG Dresden.
# open an image file
multichannel_image = imread("../../data/zfish_eye.tif")
# extract a channel
single_channel_image = multichannel_image[:,:,0]
cropped_image = single_channel_image[200:600, 500:900]
stackview.insight(cropped_image)
|
|
|
Bildfilterung#
Ein üblicher Schritt bei der Arbeit mit Fluoreszenz-Mikroskopiebildern ist das Subtrahieren der Hintergrundintensität, z.B. resultierend aus unfokussiertem Licht. Dies kann die Bildsegmentierungsergebnisse weiter unten im Workflow verbessern.
# subtract background using a top-hat filter
background_subtracted_image = cle.top_hat_box(cropped_image, radius_x=20, radius_y=20)
stackview.insight(background_subtracted_image)
|
|
|
Bildsegmentierung#
Für die Segmentierung der Zellkerne im gegebenen Bild existiert eine enorme Anzahl von Algorithmen. Hier verwenden wir einen klassischen Ansatz namens Voronoi-Otsu-Labeling, der sicherlich nicht perfekt ist.
label_image = np.asarray(cle.voronoi_otsu_labeling(background_subtracted_image, spot_sigma=4))
# show result
stackview.insight(label_image)
|
|
|
Messungen und Merkmalsextraktion#
Nachdem das Bild segmentiert ist, können wir Eigenschaften der einzelnen Objekte messen. Diese Eigenschaften sind typischerweise beschreibende statistische Parameter, die als Merkmale bezeichnet werden. Wenn wir Messungen wie die Fläche oder die mittlere Intensität ableiten, extrahieren wir diese beiden Merkmale.
statistics = measure.regionprops_table(label_image,
intensity_image=cropped_image,
properties=('area', 'mean_intensity', 'major_axis_length', 'minor_axis_length'))
Arbeiten mit Tabellen#
Das oben erstellte statistics-Objekt enthält eine Python-Datenstruktur, ein Wörterbuch von Messvektoren, was nicht am intuitivsten zu betrachten ist. Daher konvertieren wir es in eine Tabelle. Datenwissenschaftler nennen diese Tabellen oft DataFrames, die in der pandas-Bibliothek verfügbar sind.
dataframe = pd.DataFrame(statistics)
Wir können bestehende Tabellenspalten verwenden, um andere Messungen zu berechnen, wie zum Beispiel das aspect_ratio.
dataframe['aspect_ratio'] = dataframe['major_axis_length'] / dataframe['minor_axis_length']
dataframe
| area | mean_intensity | major_axis_length | minor_axis_length | aspect_ratio | |
|---|---|---|---|---|---|
| 0 | 294.0 | 36604.625850 | 25.656180 | 18.800641 | 1.364644 |
| 1 | 91.0 | 37379.769231 | 20.821990 | 6.053507 | 3.439658 |
| 2 | 246.0 | 44895.308943 | 21.830827 | 14.916032 | 1.463581 |
| 3 | 574.0 | 44394.637631 | 37.788705 | 19.624761 | 1.925563 |
| 4 | 518.0 | 45408.903475 | 26.917447 | 24.872908 | 1.082199 |
| ... | ... | ... | ... | ... | ... |
| 108 | 568.0 | 48606.121479 | 37.357606 | 19.808121 | 1.885974 |
| 109 | 175.0 | 25552.074286 | 17.419031 | 13.675910 | 1.273702 |
| 110 | 460.0 | 39031.419565 | 26.138592 | 23.522578 | 1.111213 |
| 111 | 407.0 | 39343.292383 | 28.544027 | 19.563792 | 1.459023 |
| 112 | 31.0 | 29131.322581 | 6.892028 | 5.711085 | 1.206781 |
113 rows × 5 columns
Plotten#
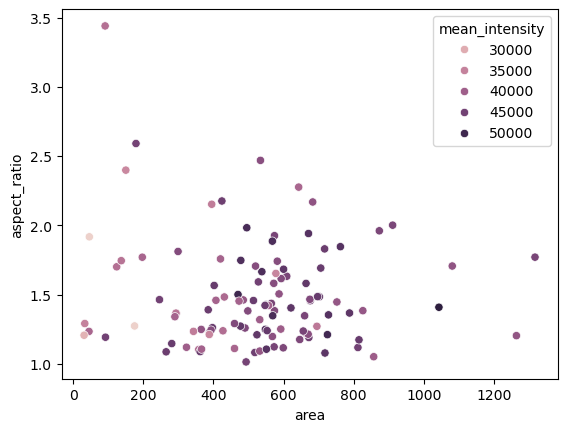
Messungen können mithilfe von Diagrammen visualisiert werden.
seaborn.scatterplot(dataframe, x='area', y='aspect_ratio', hue='mean_intensity')
<Axes: xlabel='area', ylabel='aspect_ratio'>
Deskriptive Statistik#
Ausgehend von dieser Tabelle können wir Statistiken verwenden, um einen Überblick über die gemessenen Daten zu erhalten.
mean_area = np.mean(dataframe['area'])
stddev_area = np.std(dataframe['area'])
print("Die durchschnittliche Zellkernfläche beträgt", mean_area, "+-", stddev_area, "Pixel")
Mean nucleus area is 524.4247787610619 +- 231.74703195433014 pixels
Klassifizierung#
Für ein besseres Verständnis der internen Struktur von Geweben, aber auch zur Korrektur von Artefakten in Bildverarbeitungs-Workflows, können wir Zellen klassifizieren, zum Beispiel nach ihrer Größe und Form.
object_classifier = apoc.ObjectClassifier('../../data/blobs_classifier.cl')
classification_image = object_classifier.predict(label_image, cropped_image)
stackview.imshow(classification_image)

