Gauss-Otsu-Markierung#
Bisher haben wir Bilder mit Filtern wie der Gauß-Unschärfe verarbeitet, Schwellenwert-Algorithmen wie die Otsu-Methode angewandt und Objekte mithilfe der Markierung zusammenhängender Komponenten gekennzeichnet. Die Kombination dieser drei Algorithmen ist so häufig, dass es Komfortfunktionen gibt, um diese drei Operationen in einem Durchgang auszuführen. Dieser Arbeitsablauf zur Bildsegmentierung ist ein sehr einfacher Ansatz, z.B. zur Erkennung und Segmentierung von Zellkernen in Fluoreszenzmikroskopiebildern. Er funktioniert, wenn die Objekte nicht zu dicht beieinander liegen.
from skimage.io import imread
import matplotlib.pyplot as plt
import pyclesperanto_prototype as cle
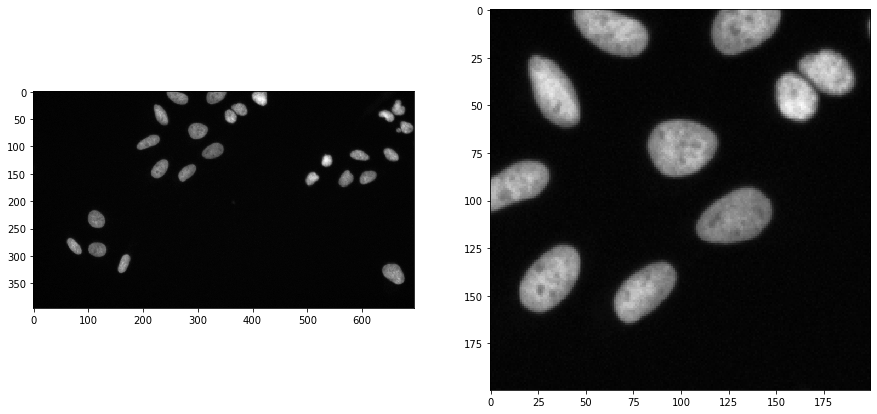
Um den Arbeitsablauf zu demonstrieren, verwenden wir Bilddaten aus der Broad Bio Image Challenge: Wir haben den Bildsatz BBBC022v1 Gustafsdottir et al., PLOS ONE, 2013 verwendet, der aus der Broad Bioimage Benchmark Collection Ljosa et al., Nature Methods, 2012 stammt.
Wir laden das Bild und schneiden zu Visualisierungszwecken einen Teilbereich aus:
input_image = imread("../../data/BBBC022/IXMtest_A02_s9.tif")[:,:,0]
input_crop = input_image[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(input_image, plot=axs[0])
cle.imshow(input_crop, plot=axs[1])
Anwendung des Algorithmus#
Gauss-Otsu-Markierung ist ein Befehl in pyclesperanto und dem skriptfähigen napari-Plugin napari-segment-blobs-and-things-with-membranes. Die Operation hat einen outline_sigma-Parameter zur Steuerung der Präzision der Umrisse der segmentierten Objekte.
sigma_outline = 1
segmented = cle.gauss_otsu_labeling(input_image, outline_sigma=sigma_outline)
segmented_crop = segmented[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(segmented, labels=True, plot=axs[0])
cle.imshow(segmented_crop, labels=True, plot=axs[1])

sigma_outline = 7
segmented = cle.gauss_otsu_labeling(input_image, outline_sigma=sigma_outline)
segmented_crop = segmented[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(segmented, labels=True, plot=axs[0])
cle.imshow(segmented_crop, labels=True, plot=axs[1])

Wie Sie sehen können, werden Objekte, die zu nahe beieinander liegen, zusammen segmentiert. In diesen Fällen wird empfohlen, Voronoi-Otsu-Labeling zu versuchen.
