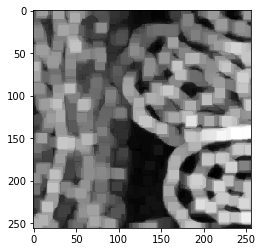
Kantenerkennung#
In clesperanto sind mehrere Filter zur Kantenerkennung implementiert.
Siehe auch
import pyclesperanto_prototype as cle
from skimage.io import imread
import matplotlib.pyplot as plt
cle.select_device("RTX")
<gfx90c on Platform: AMD Accelerated Parallel Processing (2 refs)>
blobs = imread("../../data/blobs.tif")
blobs.shape
(254, 256)
cle.imshow(blobs)
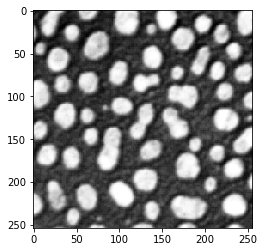
Sobel-Operator#
blobs_sobel = cle.sobel(blobs)
cle.imshow(blobs_sobel)
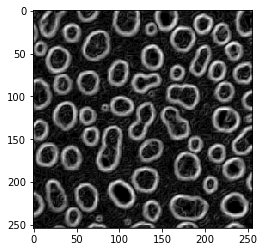
Laplace-Operator#
blobs_laplace = cle.laplace_box(blobs)
cle.imshow(blobs_laplace)

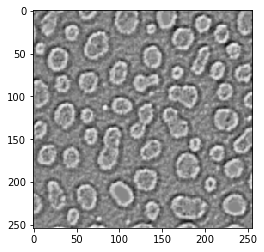
Laplacian of Gaussian#
Auch bekannt als Mexican-Hat-Filter
blobs_laplacian_of_gaussian = cle.laplace_box(cle.gaussian_blur(blobs, sigma_x=1, sigma_y=1))
cle.imshow(blobs_laplacian_of_gaussian)
blobs_laplacian_of_gaussian = cle.laplace_box(cle.gaussian_blur(blobs, sigma_x=5, sigma_y=5))
cle.imshow(blobs_laplacian_of_gaussian)

Lokaler Varianzfilter#
blobs_edges = cle.variance_box(blobs, radius_x=5, radius_y=5)
cle.imshow(blobs_edges)

Lokale Standardabweichung#
… ist einfach die Quadratwurzel der lokalen Varianz
blobs_edges = cle.standard_deviation_box(blobs, radius_x=5, radius_y=5)
cle.imshow(blobs_edges)

Kantenerkennung ist nicht Kantenverstärkung#
Intuitiv könnte man einen Kantenerkennungsfilter anwenden, um Kanten in Bildern mit Kanten zu verstärken. Versuchen wir es mit einem Bild, das Membranen zeigt. Es ist übrigens ein 3D-Bild.
image = imread("../../data/EM_C_6_c0.tif")
image.shape
(256, 256, 256)
cle.imshow(image[60])
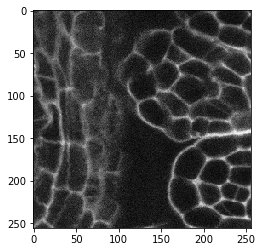
image_sobel = cle.sobel(image)
cle.imshow(image_sobel[60])
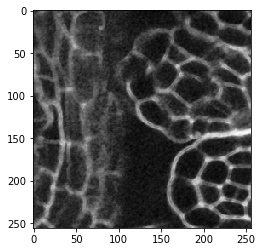
Bei genauem Hinsehen können Sie beobachten, dass die Kanten im zweiten Bild etwas dicker sind. Der Kantenerkennungsfilter erkennt zwei Kanten, die ansteigende Signalseite der Membran und die abfallende Signalseite auf der gegenüberliegenden Seite. Lassen Sie uns hineinzoomen:
fig, axs = plt.subplots(1, 2)
cle.imshow( image[60, 125:145, 135:155], plot=axs[0])
cle.imshow(cle.pull(image_sobel)[60, 125:145, 135:155], plot=axs[1])

Verstärken von Kanten#
Daher sind andere Filter nützlicher, um Kanten in einem Membranbild zu verstärken. Verstärkung kann zum Beispiel bedeuten, dass Membranen dicker werden und möglicherweise Lücken geschlossen werden.
Lokale Standardabweichung#
image_std = cle.standard_deviation_box(image, radius_x=5, radius_y=5, radius_z=5)
cle.imshow(image_std[60])

Lokales Maximum#
image_max = cle.maximum_box(image, radius_x=5, radius_y=5, radius_z=5)
cle.imshow(image_max[60])