Optimierung der Parameter für die zellbasierte Segmentierung von Membranbildern#
Workflows zur Segmentierung von Zellen aus Membranfärbungsbildern sind oft schwer zu optimieren. In diesem Notebook zeigen wir, wie man den Seeded-Watershed-Algorithmus, einen gängigen Ansatz für diese Art von Bilddaten, automatisch optimieren kann.
from napari_workflow_optimizer import JaccardLabelImageOptimizer, Workflow
from skimage.io import imread
import napari_segment_blobs_and_things_with_membranes as nsbatwm
import pyclesperanto_prototype as cle
import matplotlib.pyplot as plt
from the_segmentation_game.metrics import jaccard_index_sparse
Wir richten einen Workflow ein und fügen eine einzelne Operation ein, Seeded Watershed mit lokalen Minima als Startpunkte unter Verwendung des Napari-Plugins napari-segment-blobs-and-things-with-membranes. Der Algorithmus hat zwei Parameter: spot_sigma zur Abstimmung, wie nah Saatpunkte sein können, und outline_sigma zur Abstimmung, wie präzise die Membranen segmentiert werden sollen.
w = Workflow()
w.set("labeled", # result image name
nsbatwm.thresholded_local_minima_seeded_watershed, # operation
"input", spot_sigma=2, outline_sigma=2) # parameters
# image data source: scikit-image cells3d example, slice 28
w.set("input", imread("../../data/membranes_2d.tif"))
input_image = w.get("input")
cle.imshow(input_image)

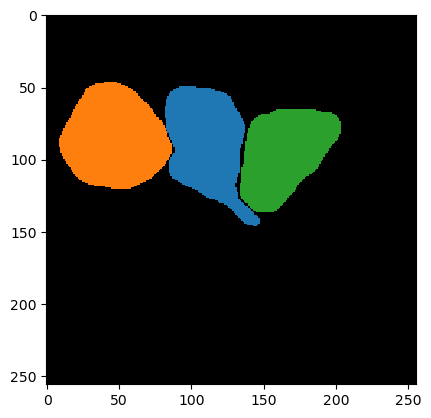
Wir erzeugen ein erstes Segmentierungsergebnis, das übersegmentiert ist, es werden offensichtlich zu viele Zellen gefunden.
result = w.get("labeled")
cle.imshow(result, labels=True)
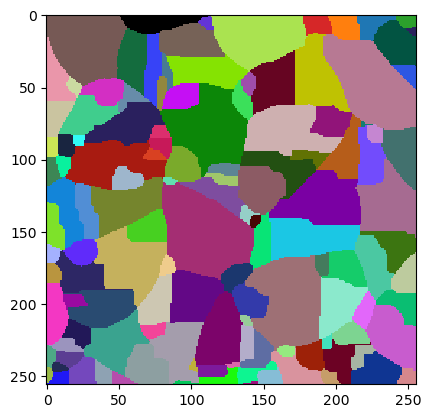
Um dem Segmentierungsalgorithmus eine Grundwahrheit zum Vergleich der Segmentierungsergebnisse zu geben, verwenden wir dieses spärlich annotierte Bild. Normalerweise reicht es aus, einige Beispielzellen genau zu annotieren. Es ist besser, Zeit in gute Segmentierungen zu investieren und nicht so viele zu zeichnen.
ground_truth = imread("../../data/membranes_2d_sparse_labels.tif")
cle.imshow(ground_truth, labels=True)
Wir können dann den JaccardLabelImageOptimizer initiieren. Nur zum Testen untersuchen wir den aktuellen Startpunkt für die Optimierung.
jlio = JaccardLabelImageOptimizer(w)
jlio.get_numeric_parameters()
[2, 2, 500]
Wir starten dann die Optimierung und geben anschließend den optimierten Parametersatz aus.
best_param = jlio.optimize("labeled", ground_truth, maxiter=100)
best_param
array([ 2.34307473, 5.6861856 , -74.78749191])
Wir können den Optimierer auch bitten, diese Parameter für uns zu setzen und das resultierende Labelbild inspizieren.
jlio.set_numeric_parameters(best_param)
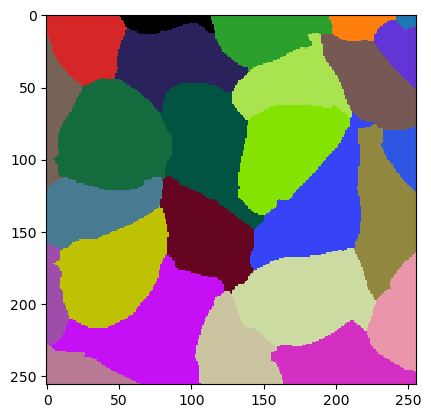
cle.imshow(w.get("labeled"), labels=True)
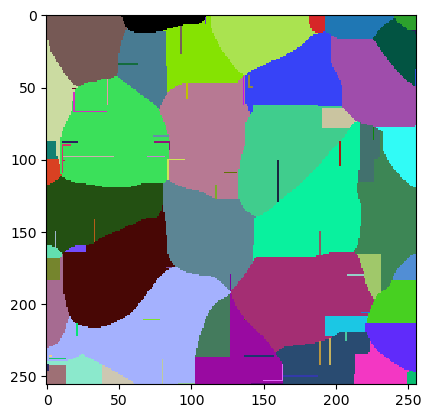
Die Qualität dieses Bildes kann durch Mittelung des Jaccard-Index der drei Grundwahrheitsobjekte gemessen werden. Die Bibliothek The Segmentation Game hat eine Funktion dafür.
jaccard_index_sparse(ground_truth, w.get("labeled"))
0.8300891581238193
Manchmal ist das Ergebnis nicht perfekt und wir möchten vielleicht einen Parameter ändern und sehen, ob das Ergebnis verbessert werden kann.
new_starting_point = best_param.copy()
new_starting_point[0] = 8
new_starting_point
array([ 8. , 5.6861856 , -74.78749191])
jlio2 = JaccardLabelImageOptimizer(w)
jlio2.set_numeric_parameters(new_starting_point)
cle.imshow(w.get("labeled"), labels=True)
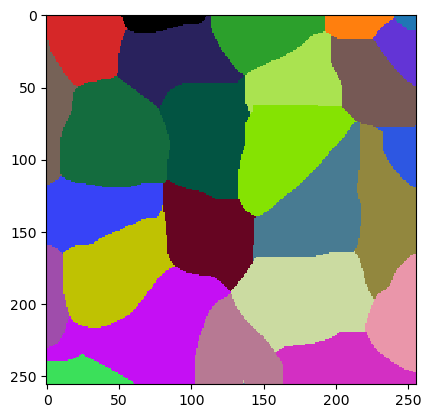
Wir können dann einen zweiten Versuch starten.
best_param = jlio.optimize("labeled", ground_truth, maxiter=100)
best_param
array([ 9.50775939, 1.39446381, -83.9132378 ])
jlio.set_numeric_parameters(best_param)
cle.imshow(w.get("labeled"), labels=True)
jaccard_index_sparse(ground_truth, w.get("labeled"))
0.8905848327576197
