Abstand zu einer Mittellinie messen#
Eine häufige Frage ist, wie man den Abstand von Punkten zum Zentrum eines segmentierten Objekts bestimmen kann. Dafür können wir das Objekt skeletonisieren, eine Abstandskarte erstellen und die Intensitäten aus der Abstandskarte an den gegebenen Punkten auslesen, um ihren Abstand zum Skelett / zur Mittellinie zu bestimmen.
Siehe auch:
https://scikit-image.org/docs/stable/auto_examples/edges/plot_skeleton.html
https://examples.itk.org/src/filtering/distancemap/maurerdistancemapofbinary/documentation
from skimage.io import imread
import napari_segment_blobs_and_things_with_membranes as nsbatwm
import napari_simpleitk_image_processing as nsitk
import numpy as np
import stackview
import pyclesperanto_prototype as cle
Ausgangspunkt: ein Binärbild#
Wir beginnen mit einem Binärbild, das wie ein Arm aussieht.
binary_arm = imread("../../data/binary_arm.tif")
stackview.insight(binary_arm)
|
|
|
Weiterhin arbeiten wir mit einer Liste von Koordinaten im X/Y-Format:
coordinates_xy = np.asarray([
[70, 80],
[70, 70],
[70, 60]]).T
Als Nächstes erstellen wir ein Labelbild, in dem die gegebenen Koordinaten markiert sind. Die erste Koordinate (Index=0 in der Liste) wird mit 1 markiert, die zweite mit 2 und so weiter. Hintergrundpixel haben den Wert 0. Wir verwenden dieses Labelbild zur Visualisierung und weiter unten werden wir es auch für die Messung verwenden.
# draw the coordinates into an image; for visualization purposes
blank_image = cle.create((binary_arm.shape))
labeled_spots = coordinate_visualization = cle.pointlist_to_labelled_spots(coordinates_xy, blank_image)
# show the labeled pixels on top of the binary image
cle.imshow(binary_arm, continue_drawing=True, max_display_intensity=1)
cle.imshow(labeled_spots, labels=True, alpha=0.6)
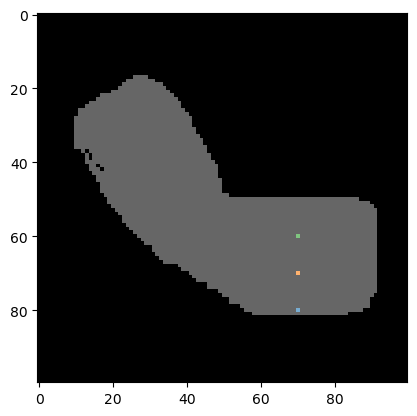
Vorverarbeitung#
Bevor wir das Bild skeletonisieren können, müssen wir die schwarzen Löcher im weißen Bereich füllen.
filled_holes = nsitk.binary_fill_holes(binary_arm)
filled_holes
|
|
n-sitk made image
|
Skeletonisierung#
Das Skelett eines Binärbildes ist eine dünne Linie in der Mitte der weißen Bereiche.
skeleton = nsbatwm.skeletonize(filled_holes)
skeleton
<__array_function__ internals>:200: RuntimeWarning: Converting input from bool to <class 'numpy.uint8'> for compatibility.
|
|
nsbatwm made image
|
Abstandskarte#
Als Nächstes erstellen wir eine Signed Maurer Distance Map. Abstandskarten sind Bilder, bei denen die Intensität den Abstand eines Pixels zum nächsten weißen Pixel im Binärbild darstellt, aus dem die Abstandskarte generiert wurde.
distance_map = nsitk.signed_maurer_distance_map(skeleton)
distance_map
|
|
n-sitk made image
|
Mit stackview.picker können wir mit der Maus über das Bild fahren und Intensitäten auslesen. Dies funktioniert nur in einer Jupyter-ähnlichen Umgebung.
stackview.picker(distance_map, zoom_factor=3)
Messungen#
Jetzt können wir die Intensität in der Abstandskarte an den gegebenen Positionen der markierten Punkte auslesen.
values_at_positions = cle.read_intensities_from_positions(coordinates_xy, distance_map)
np.asarray(values_at_positions)
array([[-15.033297 , -5.0990195, -4. ]], dtype=float32)
Übung#
Verwenden Sie die Abstandskarte auf dem binary_arm Bild, um den Abstand der drei Punkte vom Rand des Arms zu bestimmen, anstatt von der Mittellinie.
