Bestimmung der Punktspreizfunktion aus einem Kugelbild durch Mittelung#
Um ein Mikroskopbild korrekt zu entfalten, sollten wir die Punktspreizfunktion (PSF) des Mikroskops bestimmen.
Siehe auch
import numpy as np
from skimage.io import imread, imsave
from pyclesperanto_prototype import imshow
import pyclesperanto_prototype as cle
import pandas as pd
import matplotlib.pyplot as plt
Die hier verwendeten Beispielbilddaten wurden von Bert Nitzsche und Robert Haase (beide damals am MPI-CBG) in der Light Microscopy Facility des MPI-CBG aufgenommen. Der Vollständigkeit halber beträgt die Voxelgröße 0,022x0,022x0,125 µm^3.
bead_image = imread('../../data/Bead_Image1_crop.tif')
bead_image.shape
(41, 150, 150)
Unser Beispielbild zeigt fluoreszierende Kugeln, idealerweise mit einem Durchmesser, der kleiner ist als die Auflösung des Bildgebungssystems. Darüber hinaus sollten die Kugeln Licht in der gleichen Wellenlänge emittieren wie die Probe, die wir später entfalten möchten. Im folgenden Bildausschnitt sehen wir vier fluoreszierende Kugeln. Es wird empfohlen, ein größeres Sichtfeld mit mindestens 25 Kugeln aufzunehmen. Stellen Sie auch sicher, dass die Kugeln nicht aneinander kleben und spärlich verteilt sind.
imshow(cle.maximum_x_projection(bead_image), colorbar=True)
imshow(cle.maximum_y_projection(bead_image), colorbar=True)
imshow(cle.maximum_z_projection(bead_image), colorbar=True)



Um eine durchschnittliche PSF zu bestimmen, können wir technisch gesehen alle einzelnen Kugeln ausschneiden, sie ausrichten und dann die Bilder mitteln. Daher segmentieren wir die Objekte und bestimmen ihren Massenschwerpunkt.
# Objekte segmentieren
label_image = cle.voronoi_otsu_labeling(bead_image)
imshow(label_image, labels=True)
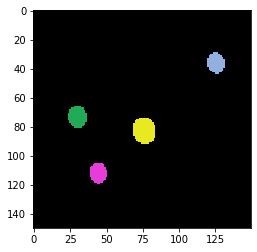
# Massenschwerpunkt für jedes Objekt bestimmen
stats = cle.statistics_of_labelled_pixels(bead_image, label_image)
df = pd.DataFrame(stats)
df[["mass_center_x", "mass_center_y", "mass_center_z"]]
| mass_center_x | mass_center_y | mass_center_z | |
|---|---|---|---|
| 0 | 30.107895 | 73.028938 | 23.327475 |
| 1 | 44.293156 | 111.633430 | 23.329062 |
| 2 | 76.092850 | 82.453033 | 23.299677 |
| 3 | 125.439606 | 35.972496 | 23.390951 |
PSF-Mittelung#
Als nächstes werden wir über die Kugeln iterieren und sie ausschneiden, indem wir sie in ein kleineres PSF-Bild übersetzen.
# Größe des zukünftigen PSF-Bildes konfigurieren
psf_radius = 20
size = psf_radius * 2 + 1
# PSF initialisieren
single_psf_image = cle.create([size, size, size])
avg_psf_image = cle.create([size, size, size])
num_psfs = len(df)
for index, row in df.iterrows():
x = row["mass_center_x"]
y = row["mass_center_y"]
z = row["mass_center_z"]
print("Kugel", index, "an Position", x, y, z)
# PSF in die richtige Position in einem kleineren Bild verschieben
cle.translate(bead_image, single_psf_image,
translate_x= -x + psf_radius,
translate_y= -y + psf_radius,
translate_z= -z + psf_radius)
# visualisieren
fig, axs = plt.subplots(1,3)
imshow(cle.maximum_x_projection(single_psf_image), plot=axs[0])
imshow(cle.maximum_y_projection(single_psf_image), plot=axs[1])
imshow(cle.maximum_z_projection(single_psf_image), plot=axs[2])
# mitteln
avg_psf_image = avg_psf_image + single_psf_image / num_psfs
Bead 0 at position 30.107894897460938 73.02893829345703 23.32747459411621
Bead 1 at position 44.293155670166016 111.63343048095703 23.32906150817871
Bead 2 at position 76.09284973144531 82.45303344726562 23.2996768951416
Bead 3 at position 125.43960571289062 35.972496032714844 23.39095115661621



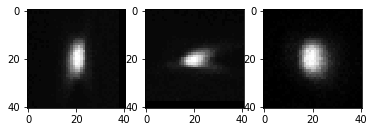
Die durchschnittliche PSF sieht dann so aus:
fig, axs = plt.subplots(1,3)
imshow(cle.maximum_x_projection(avg_psf_image), plot=axs[0])
imshow(cle.maximum_y_projection(avg_psf_image), plot=axs[1])
imshow(cle.maximum_z_projection(avg_psf_image), plot=axs[2])

avg_psf_image.min(), avg_psf_image.max()
(0.0, 94.5)
Nachdem wir eine gut zentrierte PSF bestimmt haben, können wir sie für die spätere Wiederverwendung speichern. Bevor wir das tun, normalisieren wir die PSF. Ziel ist es, ein Bild zu haben, bei dem die Gesamtintensität 1 beträgt. Dies stellt sicher, dass ein Bild, das später mit dieser PSF entfaltet wird, den Intensitätsbereich des Bildes nicht verändert.
normalized_psf = avg_psf_image / np.sum(avg_psf_image)
imshow(normalized_psf, colorbar=True)

normalized_psf.min(), normalized_psf.max()
(0.0, 0.0006259646)
imsave('../../data/psf.tif', normalized_psf)
C:\Users\rober\AppData\Local\Temp\ipykernel_16716\3265681491.py:1: UserWarning: ../../data/psf.tif is a low contrast image
imsave('../../data/psf.tif', normalized_psf)
