Intensitätsmessung an Labelgrenzen#
In einigen Anwendungen ist es sinnvoll, die Intensität an Labelgrenzen zu messen. Zum Beispiel kann man zur Messung der Signalintensität in einem Bild, das die Kernhülle zeigt, Zellkerne segmentieren, ihre Grenzen identifizieren und dann dort die Intensität messen.
import numpy as np
from skimage.io import imread, imshow
import pyclesperanto_prototype as cle
from cellpose import models, io
from skimage import measure
import matplotlib.pyplot as plt
2022-02-24 10:05:10,405 [INFO] WRITING LOG OUTPUT TO C:\Users\rober\.cellpose\run.log
Der Beispieldatensatz#
In diesem Beispiel laden wir ein Bild, das ein Zebrafischauge zeigt, mit freundlicher Genehmigung von Mauricio Rocha Martins, Norden-Labor, MPI CBG Dresden.
multichannel_image = imread("../../data/zfish_eye.tif")
multichannel_image.shape
(1024, 1024, 3)
cropped_image = multichannel_image[200:600, 500:900]
nuclei_channel = cropped_image[:,:,0]
cle.imshow(nuclei_channel)
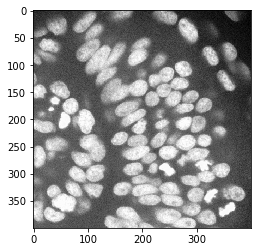
Bildsegmentierung#
Zunächst verwenden wir Cellpose, um die Zellen zu segmentieren
# load cellpose model
model = models.Cellpose(gpu=False, model_type='nuclei')
# apply model
channels = [0,0] # This means we are processing single channel greyscale images.
label_image, flows, styles, diams = model.eval(nuclei_channel, diameter=None, channels=channels)
# show result
cle.imshow(label_image, labels=True)
2022-02-24 10:05:10,623 [INFO] >>>> using CPU
2022-02-24 10:05:10,675 [INFO] ~~~ ESTIMATING CELL DIAMETER(S) ~~~
2022-02-24 10:05:13,858 [INFO] estimated cell diameter(s) in 3.18 sec
2022-02-24 10:05:13,859 [INFO] >>> diameter(s) =
2022-02-24 10:05:13,860 [INFO] [ 29.64 ]
2022-02-24 10:05:13,860 [INFO] ~~~ FINDING MASKS ~~~
2022-02-24 10:05:18,649 [INFO] >>>> TOTAL TIME 7.97 sec
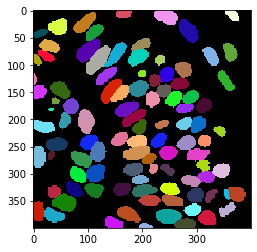
Markierung von Pixeln an Labelgrenzen#
Als Nächstes extrahieren wir den Umriss der segmentierten Zellkerne.
binary_borders = cle.detect_label_edges(label_image)
labeled_borders = binary_borders * label_image
cle.imshow(label_image, labels=True)
cle.imshow(binary_borders)
cle.imshow(labeled_borders, labels=True)

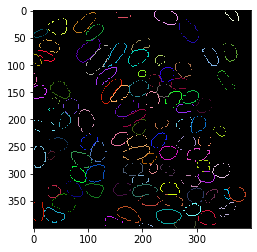
Erweiterung der Umrisse#
Wir erweitern die Umrisse ein wenig, um eine robustere Messung zu erhalten.
extended_outlines = cle.dilate_labels(labeled_borders, radius=2)
cle.imshow(extended_outlines, labels=True)
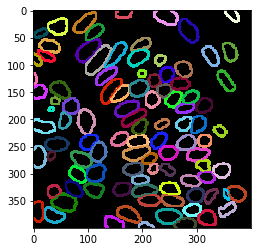
Überlagerungsvisualisierung#
Mit diesem Labelbild der Zellkernumrisse können wir die Intensität in der Kernhülle messen.
nuclear_envelope_channel = cropped_image[:,:,2]
cle.imshow(nuclear_envelope_channel)
cle.imshow(nuclear_envelope_channel, alpha=0.5, continue_drawing=True)
cle.imshow(extended_outlines, alpha=0.5, labels=True)

Labelintensitätsstatistiken#
Die Messung der Intensität im Bild funktioniert mit den richtigen Intensitäts- und Labelbildern.
stats = cle.statistics_of_labelled_pixels(nuclear_envelope_channel, extended_outlines)
stats["mean_intensity"]
array([35529.4 , 32835.07 , 36713.887, 37146.348, 49462.39 , 36392.6 ,
37998.375, 48974.945, 31805.87 , 50451.793, 41006.047, 50854.016,
36167.547, 41332.32 , 37815.766, 35121.38 , 43859.945, 40292.875,
31583.992, 38933.57 , 32297.547, 39140.766, 37072.31 , 45990.57 ,
39800.613, 37804.99 , 39092.43 , 39510.848, 40534.81 , 42057.293,
44815.844, 42855.754, 38408.246, 41257.594, 37996.895, 38568.465,
42331.266, 34748.973, 44219.844, 41986.086, 38606.215, 39008.094,
36411.05 , 48155.797, 43781.97 , 38315.12 , 36048.39 , 37739.277,
46268.816, 35808.32 , 37388.312, 37682.21 , 42932.72 , 38168.293,
40489.73 , 43073.066, 40973.285, 40975.246, 39292.848, 38555.766,
38219.785, 40054.242, 37356.87 , 45014.8 , 37211.668, 47025.47 ,
30218.678, 33988.027, 37338.41 , 38500.85 , 38546.777, 40611.742,
40391.453, 41024.46 , 37840.246, 41342.793, 39329.625, 43311.016,
37829.074, 39949.82 , 39316.496, 40966.48 , 34066.7 , 34929.863,
40356.445, 31959.607, 39480.855, 39194.027, 46274.582, 31316.648,
37623.61 , 40962.016, 39203.37 , 45368.703, 37830.832, 35296.93 ,
37756.1 , 39108.93 , 40739.543], dtype=float32)
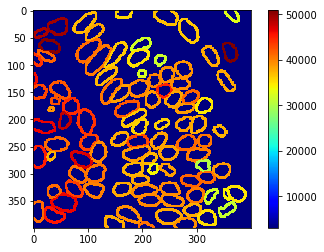
Parametrische Karten#
Diese Messungen können auch mithilfe von parametrischen Karten visualisiert werden
intensity_map = cle.mean_intensity_map(nuclear_envelope_channel, extended_outlines)
cle.imshow(intensity_map, min_display_intensity=3000, colorbar=True, colormap="jet")
Übung#
Messen und visualisieren Sie die Intensität an den Labelgrenzen im Zellkernkanal.
