Mehrdimensionale Bildstapel#
Mehrdimensionale Bilddaten können auf ähnliche Weise wie Mehrkanal-Bilddaten gehandhabt werden.
Dreidimensionale Bildstapel#
Es gibt auch Bilder mit drei räumlichen Dimensionen: X, Y und Z. Typische Beispiele finden Sie in der Mikroskopie und in der medizinischen Bildgebung. Werfen wir einen Blick auf einen Magnetresonanztomographie (MRT) Datensatz:
from skimage.io import imread
from pyclesperanto_prototype import imshow
image_stack = imread('../../data/Haase_MRT_tfl3d1.tif')
image_stack.shape
(192, 256, 256)
Wir sehen, dass die Daten tatsächlich drei Dimensionen haben, in diesem Fall 192 Z-Ebenen und 256 X- und Y-Pixel. Wir können sie mit pyclesperantos imshow anzeigen:
imshow(image_stack)
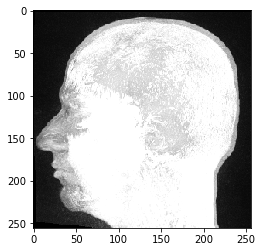
Dieser MRT-Datensatz sieht ungewöhnlich aus, weil wir eine Maximumintensitätsprojektion betrachten, die pyclesperantos Standardmethode zur Visualisierung dreidimensionaler Daten ist.
Bildschnitte#
Wir können einzelne Bildschnitte untersuchen, indem wir ihren Index in unserem 3D-Numpy-Array angeben und diesmal Matplotlibs imshow zur Visualisierung verwenden:
import matplotlib.pyplot as plt
fig, axs = plt.subplots(1, 3, figsize=(15,15))
# zeige drei planare Bilder
axs[0].imshow(image_stack[48], cmap='Greys_r')
axs[1].imshow(image_stack[96], cmap='Greys_r')
axs[2].imshow(image_stack[144], cmap='Greys_r');
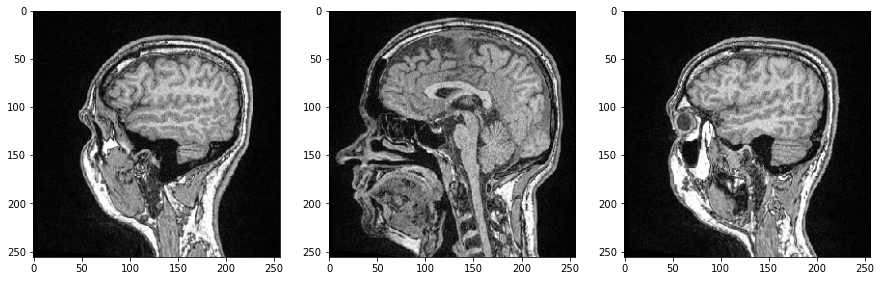
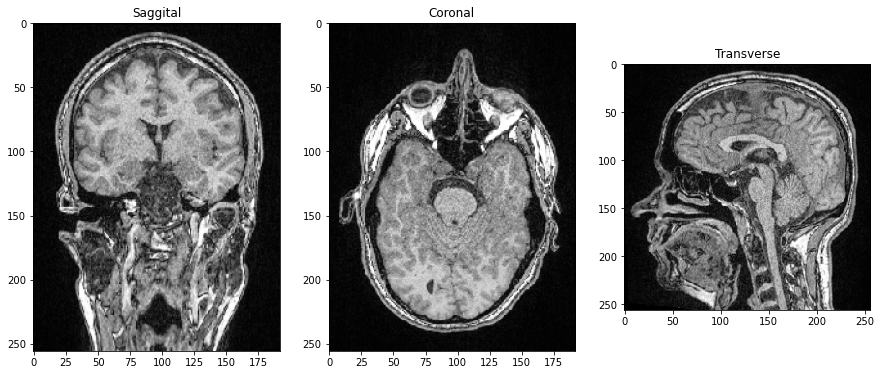
Da alle drei Dimensionen räumliche Dimensionen sind, können wir auch Schnitte orthogonal zur Bildebene und entsprechend den Anatomischen Ebenen erstellen. Um die Bilder korrekt auszurichten, können wir ihre Achsen transponieren, indem wir am Ende .T hinzufügen.
saggital = image_stack[:,:,128].T
coronal = image_stack[:,128,:].T
transverse = image_stack[96]
fig, axs = plt.subplots(1, 3, figsize=(15,15))
# zeige orthogonale Ebenen
axs[0].imshow(saggital, cmap='Greys_r')
axs[0].set_title('Sagittal')
axs[1].imshow(coronal, cmap='Greys_r')
axs[1].set_title('Koronal')
axs[2].imshow(transverse, cmap='Greys_r')
axs[2].set_title('Transversal');
Videos#
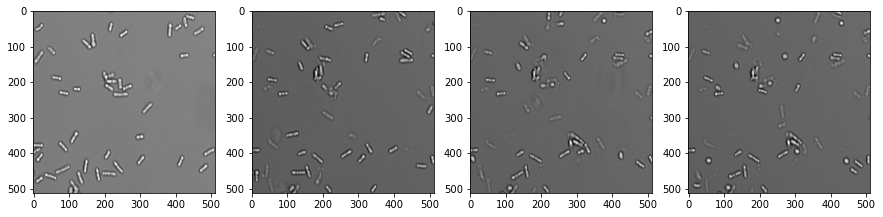
Wenn ein Bilddatensatz eine zeitliche Dimension hat, nennen wir ihn ein Video. Die Verarbeitung von Videos funktioniert ähnlich wie bei Mehrkanal-Bildern und Bildstapeln. Öffnen wir einen Mikroskopie-Datensatz, der Hefezellen zeigt, die sich im Laufe der Zeit abrunden. (Bilddaten mit freundlicher Genehmigung von Anne Esslinger, Alberti-Labor, MPI CBG)
video = imread('../../data/rounding_assay.tif')
video.shape
(64, 512, 512)
fig, axs = plt.subplots(1, 4, figsize=(15,15))
# zeige drei planare Bilder
axs[0].imshow(video[0], cmap='Greys_r')
axs[1].imshow(video[5], cmap='Greys_r')
axs[2].imshow(video[10], cmap='Greys_r')
axs[3].imshow(video[15], cmap='Greys_r');
n-dimensionale Daten#
Hochdimensionale Daten sind in der Mikroskopie ziemlich häufig. Um sie korrekt zu verarbeiten, muss man sorgfältig untersuchen, welche Dimensionen ein Bilddatensatz hat. Wir können die Möglichkeiten anhand des mitosis-Datensatzes erkunden:
mitosis = imread('../../data/mitosis.tif')
mitosis.shape
(51, 5, 2, 196, 171)
Tipp: Öffnen Sie den Datensatz in ImageJ/Fiji, um zu verstehen, wofür diese Zahlen stehen. Sie können dort sehen, dass der Mitose-Datensatz
51 Frames,
5 Z-Schnitte,
2 Kanäle und
eine Größe von 171 x 196 Pixeln hat.
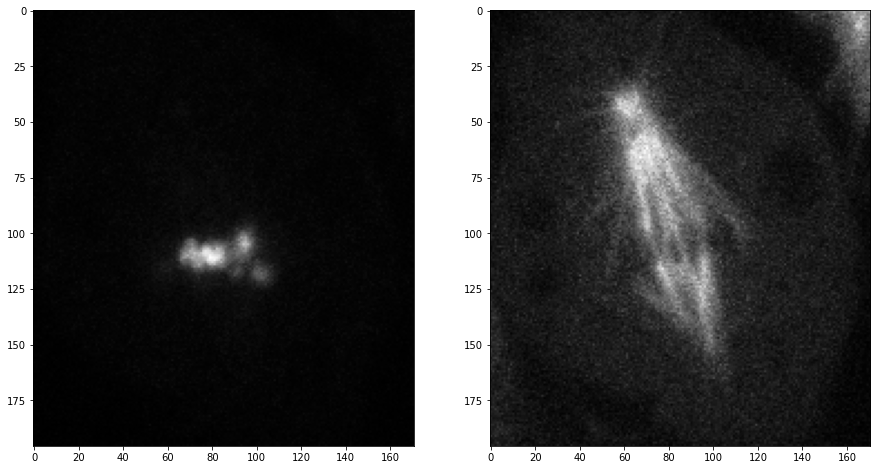
Wir greifen jetzt die Kanäle 1 und 2 des ersten Zeitpunkts (Index 0) in der mittleren Ebene (Index 2) heraus:
timepoint = 0
plane = 2
channel1 = mitosis[timepoint, plane, 0]
channel2 = mitosis[timepoint, plane, 1]
fig, axs = plt.subplots(1, 2, figsize=(15,15))
axs[0].imshow(channel1, cmap='Greys_r')
axs[1].imshow(channel2, cmap='Greys_r');
Übung#
Öffnen Sie den Mitose-Datensatz, wählen Sie drei Zeitpunkte aus und zeigen Sie sie nebeneinander. Die resultierende Abbildung sollte drei Spalten und zwei Zeilen haben. In der ersten Zeile wird Kanal 1 angezeigt und der zweite Kanal darunter.
