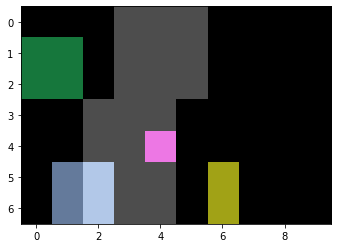Messung von Abständen zwischen Objekten#
Dieses Notebook zeigt, wie man den Abstand von Objekten in einem Bild zum nächstgelegenen Objekt in einem anderen Bild messen kann. Ein Anwendungsfall dafür ist die Messung des Abstands von Objekten wie Zellen zu einem Gefäß. Das folgende Verfahren wird in 2D demonstriert und sollte analog in 3D funktionieren.
import numpy as np
import pyclesperanto_prototype as cle
import napari_simpleitk_image_processing as nsitk
import pandas as pd
Als Beispieldaten verwenden wir zwei synthetische Bilder: ein Binärbild, das zeigt, wo sich ein Gefäß als Streifen befindet, und ein Labelbild, das zeigt, wo sich Zellen / Objekte befinden.
vessel = np.asarray([
[0, 0, 0, 1, 1, 1, 0, 0, 0, 0],
[0, 0, 0, 1, 1, 1, 0, 0, 0, 0],
[0, 0, 0, 1, 1, 1, 0, 0, 0, 0],
[0, 0, 1, 1, 1, 0, 0, 0, 0, 0],
[0, 0, 1, 1, 1, 0, 0, 0, 0, 0],
[0, 0, 1, 1, 1, 0, 0, 0, 0, 0],
[0, 0, 1, 1, 1, 0, 0, 0, 0, 0],
])
objects = np.asarray([
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[1, 1, 0, 0, 0, 0, 0, 0, 0, 0],
[1, 1, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 5, 5],
[0, 0, 0, 0, 2, 0, 0, 0, 5, 5],
[0, 4, 4, 0, 0, 0, 3, 0, 0, 0],
[0, 4, 4, 0, 0, 0, 3, 0, 0, 0],
])
cle.imshow(vessel, continue_drawing=True)
cle.imshow(objects, labels=True, alpha=0.7)

Abstandskarten#
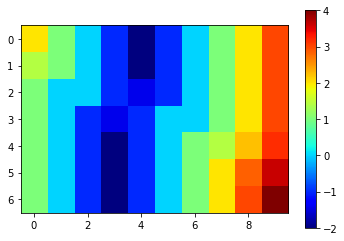
Zur Messung des durchschnittlichen Abstands markierter Pixel zu einem anderen Objekt beinhaltet eine Strategie die Verwendung von Abstandskarten. Hier verwenden wir die in SimpleITK implementierte signierte Maurer-Abstandskarte. Genauer gesagt verwenden wir das skriptfähige napari-Plugin napari-simpleitk-image-processing, um auf diese Karte zuzugreifen. Wenn die Werte positiv sind, befinden wir uns außerhalb des Gefäßes. Negative Werte deuten darauf hin, dass wir uns im Inneren befinden.
not_vessel = cle.binary_not(vessel)
distance_from_vessel = nsitk.signed_maurer_distance_map(not_vessel)
cle.imshow(distance_from_vessel, colorbar=True, colormap="jet")
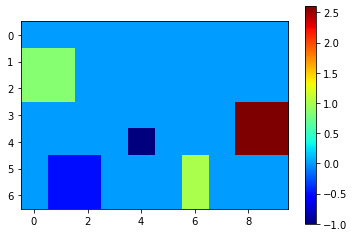
Objektabstandskarte#
Als Nächstes messen wir die durchschnittliche Intensität in der Abstandskarte, wo sich die Objekte befinden. Diese Intensitäten repräsentieren den durchschnittlichen Abstand dieser Objekte zum Gefäß. Die Hintergrundintensität in diesen Karten (in unserem Fall der Abstand) ist definitionsgemäß 0.
mean_distance_map = cle.mean_intensity_map(distance_from_vessel, objects)
cle.imshow(mean_distance_map, colorbar=True, colormap="jet")
Alternativ zu dieser Visualisierung können wir den durchschnittlichen Abstand auch mithilfe von Pixelstatistiken auslesen und mit einem pandas Dataframe ausgeben.
statistics = cle.statistics_of_labelled_pixels(distance_from_vessel, objects)
table = pd.DataFrame(statistics)
table = table[['label', 'mean_intensity']]
table = table.rename(columns={'mean_intensity':'mean_distance'})
table
| label | mean_distance | |
|---|---|---|
| 0 | 1 | 0.853553 |
| 1 | 2 | -1.000000 |
| 2 | 3 | 1.000000 |
| 3 | 4 | -0.500000 |
| 4 | 5 | 2.599586 |
Filtern von Objekten#
Sie können auch Objekte nach ihrem Abstand zum Gefäß filtern.
min_distance = -4
max_distance = 2
objects_close_by_vessel = cle.exclude_labels_with_map_values_out_of_range(
mean_distance_map,
objects,
minimum_value_range=min_distance,
maximum_value_range=max_distance
)
cle.imshow(vessel, continue_drawing=True)
cle.imshow(objects_close_by_vessel, labels=True, alpha=0.7)