Vergleich von Messungen aus verschiedenen Bibliotheken#
Wir vergleichen nun die Implementierung der Messung perimeter in napari-skimage-regionprops regionprops_table und napari-simpleitk-image-processing label_statistics.
import numpy as np
import pandas as pd
from skimage.io import imread
from pyclesperanto_prototype import imshow
from skimage import filters
from skimage import measure
import matplotlib.pyplot as plt
from napari_skimage_regionprops import regionprops_table
from skimage import filters
from napari_simpleitk_image_processing import label_statistics
C:\Users\maral\mambaforge\envs\feature_blogpost\lib\site-packages\morphometrics\measure\label.py:7: TqdmExperimentalWarning: Using `tqdm.autonotebook.tqdm` in notebook mode. Use `tqdm.tqdm` instead to force console mode (e.g. in jupyter console)
from tqdm.autonotebook import tqdm
Dafür benötigen wir ein Bild und ein Labelbild.
# Bild laden
image = imread("../../data/blobs.tif")
# Rauschunterdrückung
blurred_image = filters.gaussian(image, sigma=1)
# Binarisierung
threshold = filters.threshold_otsu(blurred_image)
thresholded_image = blurred_image >= threshold
# Labeling
label_image = measure.label(thresholded_image)
# Visualisierung
imshow(label_image, labels=True)
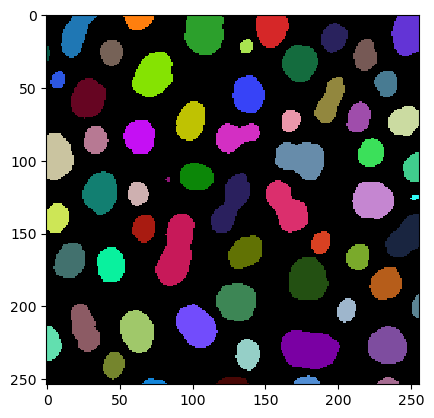
Wir leiten die Umfangsmessungen mit der Funktion regionprops_table ab
skimage_statistics = regionprops_table(image, label_image, perimeter = True)
skimage_statistics
| label | area | bbox_area | equivalent_diameter | convex_area | max_intensity | mean_intensity | min_intensity | perimeter | perimeter_crofton | standard_deviation_intensity | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 429 | 750 | 23.371345 | 479 | 232.0 | 191.440559 | 128.0 | 89.012193 | 87.070368 | 29.793138 |
| 1 | 2 | 183 | 231 | 15.264430 | 190 | 224.0 | 179.846995 | 128.0 | 53.556349 | 53.456120 | 21.270534 |
| 2 | 3 | 658 | 756 | 28.944630 | 673 | 248.0 | 205.604863 | 120.0 | 95.698485 | 93.409370 | 29.392255 |
| 3 | 4 | 433 | 529 | 23.480049 | 445 | 248.0 | 217.515012 | 120.0 | 77.455844 | 76.114262 | 35.852345 |
| 4 | 5 | 472 | 551 | 24.514670 | 486 | 248.0 | 213.033898 | 128.0 | 83.798990 | 82.127941 | 28.741080 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 57 | 58 | 213 | 285 | 16.468152 | 221 | 224.0 | 184.525822 | 120.0 | 52.284271 | 52.250114 | 28.255467 |
| 58 | 59 | 79 | 108 | 10.029253 | 84 | 248.0 | 184.810127 | 128.0 | 39.313708 | 39.953250 | 33.739912 |
| 59 | 60 | 88 | 110 | 10.585135 | 92 | 216.0 | 182.727273 | 128.0 | 45.692388 | 46.196967 | 24.417173 |
| 60 | 61 | 52 | 75 | 8.136858 | 56 | 248.0 | 189.538462 | 128.0 | 30.692388 | 32.924135 | 37.867411 |
| 61 | 62 | 48 | 68 | 7.817640 | 53 | 224.0 | 173.833333 | 128.0 | 33.071068 | 35.375614 | 27.987596 |
62 rows × 11 columns
… und mit der Funktion label_statistics
simpleitk_statistics = label_statistics(image, label_image, size = False, intensity = False, perimeter = True)
simpleitk_statistics
| label | perimeter | perimeter_on_border | perimeter_on_border_ratio | |
|---|---|---|---|---|
| 0 | 1 | 87.070368 | 16.0 | 0.183759 |
| 1 | 2 | 53.456120 | 21.0 | 0.392846 |
| 2 | 3 | 93.409370 | 23.0 | 0.246228 |
| 3 | 4 | 76.114262 | 20.0 | 0.262763 |
| 4 | 5 | 82.127941 | 40.0 | 0.487045 |
| ... | ... | ... | ... | ... |
| 57 | 58 | 52.250114 | 0.0 | 0.000000 |
| 58 | 59 | 39.953250 | 18.0 | 0.450527 |
| 59 | 60 | 46.196967 | 22.0 | 0.476222 |
| 60 | 61 | 32.924135 | 15.0 | 0.455593 |
| 61 | 62 | 35.375614 | 17.0 | 0.480557 |
62 rows × 4 columns
Für diesen Vergleich interessieren wir uns nur für die Spalte mit dem Namen perimeter. Daher wählen wir diese Spalte aus und konvertieren die enthaltenen Messungen in ein numpy-Array:
skimage_perimeter = np.asarray(skimage_statistics['perimeter'])
simpleitk_perimeter = np.asarray(simpleitk_statistics['perimeter'])
Streudiagramm#
Wenn wir nun matplotlib verwenden, um die beiden Umfangsmessungen gegeneinander zu plotten, erhalten wir folgendes Streudiagramm:
plt.plot(skimage_perimeter, simpleitk_perimeter, 'o')
[<matplotlib.lines.Line2D at 0x1912b551220>]
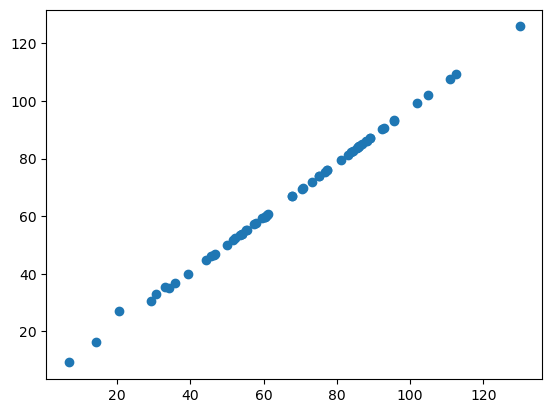
Wenn die beiden Bibliotheken den Umfang auf die gleiche Weise berechnen würden, lägen alle unsere Datenpunkte auf einer geraden Linie. Wie Sie sehen können, ist dies nicht der Fall. Dies deutet darauf hin, dass der Umfang in napari-skimage-regionprops und napari-simpleitk-image-processing unterschiedlich implementiert ist.
Bland-Altman-Diagramm#
Das Bland-Altman-Diagramm hilft dabei, den Unterschied zwischen Messungen zu visualisieren. Wir können ein Bland-Altman-Diagramm für unsere zwei verschiedenen perimeter-Implementierungen wie folgt berechnen:
# Berechne Mittelwert, Differenz, md und sd
mean = (skimage_perimeter + simpleitk_perimeter) / 2
diff = skimage_perimeter - simpleitk_perimeter
md = np.mean(diff) # Mittelwert der Differenz
sd = np.std(diff, axis = 0) # Standardabweichung der Differenz
# Füge Mittelwert und Differenz hinzu
plt.plot(mean, diff, 'o')
# Füge Linien hinzu
plt.axhline(md, color='gray', linestyle='--')
plt.axhline(md + 1.96*sd, color='gray', linestyle='--')
plt.axhline(md - 1.96*sd, color='gray', linestyle='--')
# Füge Titel und Achsenbeschriftungen hinzu
plt.title('Bland-Altman-Diagramm')
plt.xlabel('Durchschnittliche Messung')
plt.ylabel('Differenz der Messung')
Text(0, 0.5, 'Difference of Measurement')
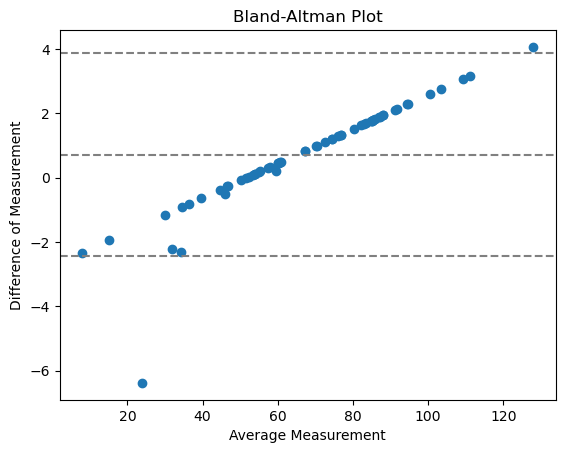
Mit
mittlere Linie = mittlere Differenz zwischen den Methoden
zwei äußere Linien = Konfidenzintervall der Übereinstimmung (CI)
Die Punkte gehen nicht gegen 0, was bedeutet, dass es keine Übereinstimmung mit zufälligem relativen Fehler gibt, sondern systematisch. Das ergibt sehr viel Sinn, da wir hier die Implementierung einer Messung vergleichen.
Übung#
Verwenden Sie die Funktionen regionprops_table und label_statistics, um feret_diameter_max Ihres Labelbildes zu messen. Erstellen Sie das Streudiagramm und das Bland-Altman-Diagramm. Denken Sie, dass feret_diameter_max in den beiden Bibliotheken unterschiedlich implementiert ist?
