(image-segmentation:voronoi-otsu-labeling=)
Voronoi-Otsu-Labeling#
Dieser Workflow zur Bildsegmentierung ist ein relativ einfacher und dennoch leistungsfähiger Ansatz, z.B. zur Erkennung und Segmentierung von Zellkernen in Fluoreszenz-Mikroskopiebildern. Ein Zellkernmarker wie Kern-GFP, DAPI oder Histon-RFP in Kombination mit verschiedenen Mikroskopietechniken kann verwendet werden, um Bilder geeigneter Art zu erzeugen.
from skimage.io import imread, imshow
import matplotlib.pyplot as plt
import pyclesperanto_prototype as cle
import napari_segment_blobs_and_things_with_membranes as nsbatwm
Um den Workflow zu demonstrieren, verwenden wir Bilddaten aus der Broad Bio Image Challenge: Wir haben den Bildsatz BBBC022v1 Gustafsdottir et al., PLOS ONE, 2013 verwendet, der aus der Broad Bioimage Benchmark Collection Ljosa et al., Nature Methods, 2012 stammt.
input_image = imread("../../data/BBBC022/IXMtest_A02_s9.tif")[:,:,0]
input_crop = input_image[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(input_image, plot=axs[0])
cle.imshow(input_crop, plot=axs[1])
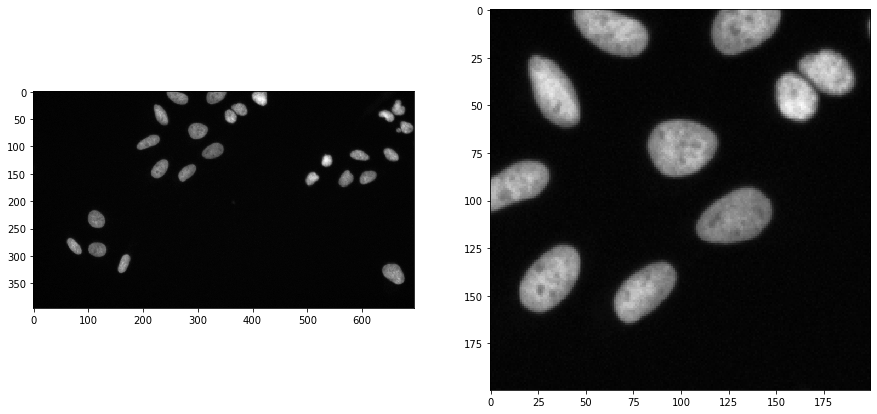
Anwendung des Algorithmus#
Voronoi-Otsu-Labeling ist ein Befehl in clesperanto, der zwei Sigma-Parameter benötigt. Das erste Sigma steuert, wie nahe die erkannten Zellen beieinander liegen können (spot_sigma), und das zweite steuert, wie genau die segmentierten Objekte umrissen werden (outline_sigma).
sigma_spot_detection = 5
sigma_outline = 1
segmented = cle.voronoi_otsu_labeling(input_image, spot_sigma=sigma_spot_detection, outline_sigma=sigma_outline)
segmented_crop = segmented[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(segmented, labels=True, plot=axs[0])
cle.imshow(segmented_crop, labels=True, plot=axs[1])
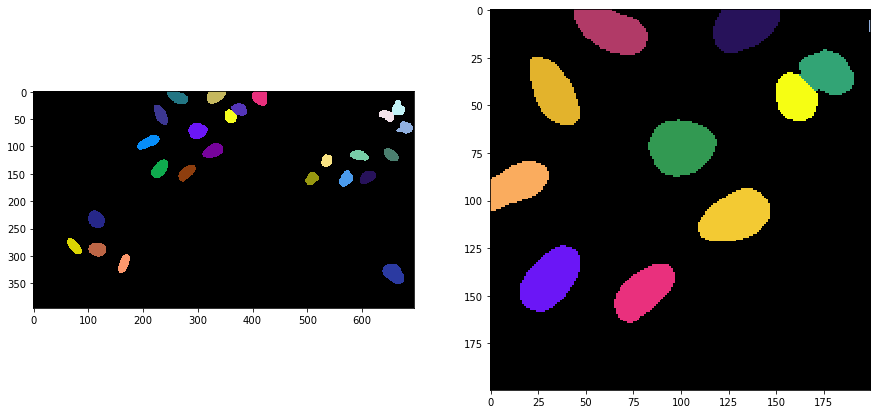
Wie funktioniert es?#
Der Voronoi-Otsu-Labeling-Workflow ist eine Kombination aus Gauß-Unschärfe, Spot-Erkennung, Schwellenwertbildung und binärer Wasserscheide. Der interessierte Leser kann den Open-Source-Code einsehen. Der Ansatz ähnelt der Anwendung einer Saatgut-Wasserscheide auf ein Binärbild, z.B. in MorphoLibJ oder scikit-image. Die Saatpunkte werden jedoch automatisch berechnet und können nicht übergeben werden.
Zu Demonstrationszwecken führen wir dies nur auf dem oben gezeigten 2D-Ausschnittbild durch. Wenn dieser Algorithmus auf 3D-Daten angewendet wird, empfiehlt es sich, diese zuerst isotrop zu machen.
image_to_segment = input_crop
print(image_to_segment.shape)
(200, 200)
Als ersten Schritt verwischen wir das Bild mit einem gegebenen Sigma und erkennen Maxima im resultierenden Bild.
blurred = cle.gaussian_blur(image_to_segment, sigma_x=sigma_spot_detection, sigma_y=sigma_spot_detection, sigma_z=sigma_spot_detection)
detected_spots = cle.detect_maxima_box(blurred, radius_x=0, radius_y=0, radius_z=0)
number_of_spots = cle.sum_of_all_pixels(detected_spots)
print("Anzahl der erkannten Spots", number_of_spots)
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(blurred, plot=axs[0])
cle.imshow(detected_spots, plot=axs[1])
number of detected spots 29.0

Weiterhin beginnen wir erneut mit dem zugeschnittenen Bild und verwischen es wieder, diesmal mit einem anderen Sigma. Anschließend wenden wir einen Schwellenwert auf das Bild an, wobei wir Otsus Schwellenwertmethode (Otsu et al 1979) verwenden.
blurred = cle.gaussian_blur(image_to_segment, sigma_x=sigma_outline, sigma_y=sigma_outline, sigma_z=sigma_outline)
binary = cle.threshold_otsu(blurred)
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(blurred, plot=axs[0])
cle.imshow(binary, plot=axs[1])

Anschließend nehmen wir das binäre Spots-Bild und das binäre Segmentierungsbild und wenden eine binary_and-Operation an, um Spots auszuschließen, die im Hintergrundbereich erkannt wurden. Diese entsprechen wahrscheinlich Rauschen.
selected_spots = cle.binary_and(binary, detected_spots)
number_of_spots = cle.sum_of_all_pixels(selected_spots)
print("Anzahl der ausgewählten Spots", number_of_spots)
fig, axs = plt.subplots(1, 3, figsize=(15, 15))
cle.imshow(detected_spots, plot=axs[0])
cle.imshow(binary, plot=axs[1])
cle.imshow(selected_spots, plot=axs[2])
number of selected spots 11.0

Als nächstes trennen wir den Bildraum zwischen den ausgewählten Spots mithilfe eines Voronoi-Diagramms, das auf die positiven Pixel im Binärbild beschränkt ist.
voronoi_diagram = cle.masked_voronoi_labeling(selected_spots, binary)
fig, axs = plt.subplots(1, 3, figsize=(15, 15))
cle.imshow(selected_spots, plot=axs[0])
cle.imshow(binary, plot=axs[1])
cle.imshow(voronoi_diagram, labels=True, plot=axs[2])

Andere Implementierungen von Voronoi-Otsu-Labeling#
Es gibt eine alternative Implementierung des Algorithmus im skriptfähigen napari-Plugin napari-segment-blobs-and-things-with-membranes.
Der Code hier ist fast identisch mit dem Code oben. Der Hauptunterschied besteht darin, dass wir nsbatwm.voronoi_otsu_labeling() anstelle von cle.voronoi_otsu_labeling() aufrufen.
sigma_spot_detection = 5
sigma_outline = 1
segmented2 = nsbatwm.voronoi_otsu_labeling(input_image, spot_sigma=sigma_spot_detection, outline_sigma=sigma_outline)
segmented_crop2 = segmented2[0:200, 200:400]
fig, axs = plt.subplots(1, 2, figsize=(15, 15))
cle.imshow(segmented2, labels=True, plot=axs[0])
cle.imshow(segmented_crop2, labels=True, plot=axs[1])

