Nachbargitter in drei Dimensionen#
Die Nachbargitter, mit denen wir in diesem Kapitel gearbeitet haben, funktionieren im Allgemeinen auch in drei Dimensionen. Es ist nur schwieriger, diese zu visualisieren.
from skimage.io import imread
import pyclesperanto_prototype as cle
import matplotlib.pyplot as plt
Die hier verwendeten Bilddaten zeigen einen Ausschnitt eines sich entwickelnden Tribolium castaneum Embryos, der mittels Lichtscheibenmikroskopie von Daniela Vorkel, Myers Labor, MPI-CBG / CSBD Dresden, aufgenommen wurde.
raw_image = imread("../../data/Lund_000500_resampled-cropped.tif")
raw_image.shape
(100, 256, 256)
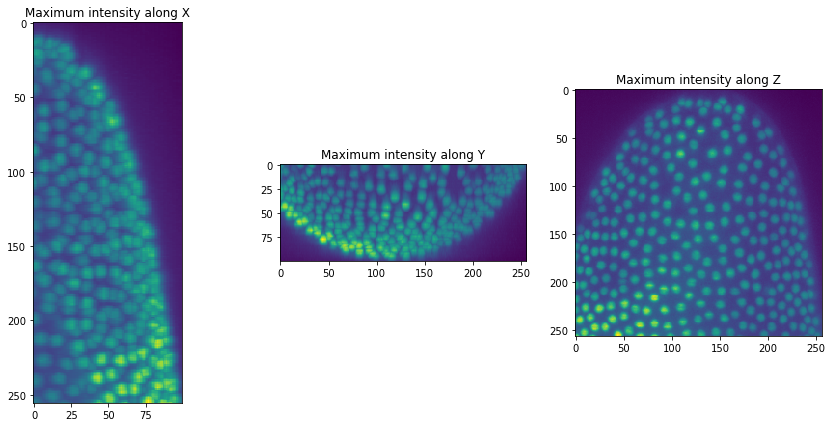
Der Einfachheit halber schreiben wir eine kurze Funktion zur Visualisierung unseres Bildstapels in drei Maximalprojektionen aus verschiedenen Perspektiven.
def orthogonal_show(image, labels=False):
fig, axs = plt.subplots(1, 3, figsize=(15, 7))
cle.imshow(cle.maximum_x_projection(image), plot=axs[0], labels=labels)
cle.imshow(cle.maximum_y_projection(image), plot=axs[1], labels=labels)
cle.imshow(cle.maximum_z_projection(image), plot=axs[2], labels=labels)
axs[0].set_title("Maximale Intensität entlang X")
axs[1].set_title("Maximale Intensität entlang Y")
axs[2].set_title("Maximale Intensität entlang Z")
orthogonal_show(raw_image)
Wir können nun Zellkerne in unserem Datensatz segmentieren.
background_subtracted = cle.top_hat_box(raw_image, radius_x=5, radius_y=5, radius_z=5)
nuclei = cle.voronoi_otsu_labeling(background_subtracted)
orthogonal_show(nuclei, labels=True)
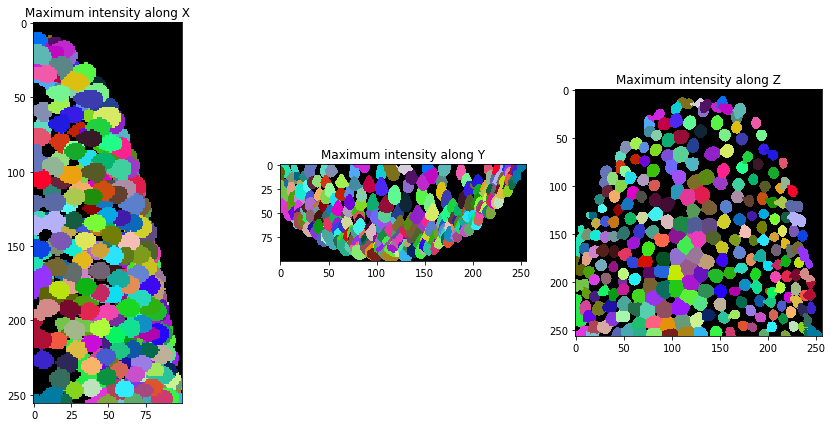
Nach der Segmentierung erweitern wir die Beschriftungen ein wenig, damit sie sich berühren.
expanded_nuclei = cle.dilate_labels(nuclei, radius=4)
orthogonal_show(expanded_nuclei, labels=True)
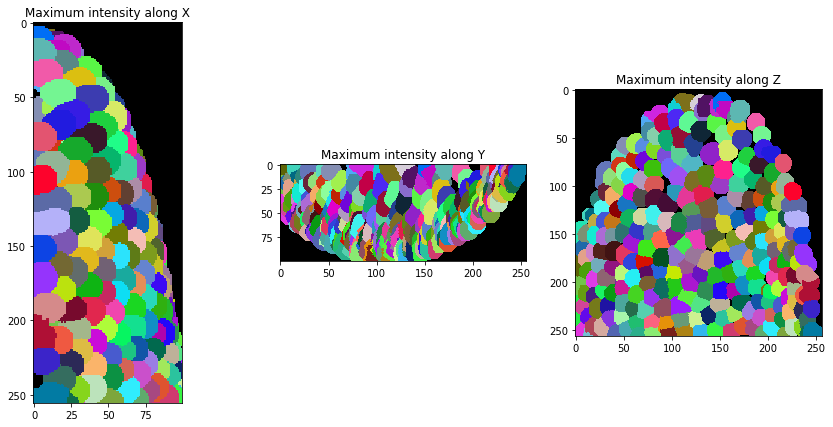
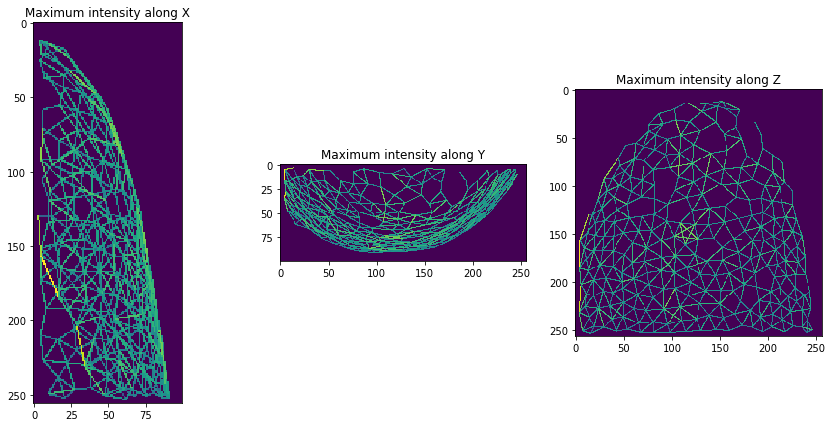
Und dann können wir ein (Schwerpunkt-)Distanznetz zwischen sich berührenden Nachbarn visualisieren.
mesh = cle.draw_distance_mesh_between_touching_labels(expanded_nuclei)
orthogonal_show(mesh)