Slicing und Zuschneiden#
Bei der Arbeit mit größeren Bilddaten ist es oft sinnvoll, Regionen auszuschneiden und sich für weitere Analysen auf diese zu konzentrieren. Zum Zuschneiden von Bildern verwenden wir die gleiche “:”-Syntax, die wir beim Indexieren in Listen und Erkunden mehrdimensionaler Bilddaten verwendet haben.
import numpy as np
from skimage.io import imread, imshow
Wir beginnen damit, ein 3D-Bild zu laden und seine Größe auszugeben.
image = imread("../../data/Haase_MRT_tfl3d1.tif")
image.shape
(192, 256, 256)
Slicing#
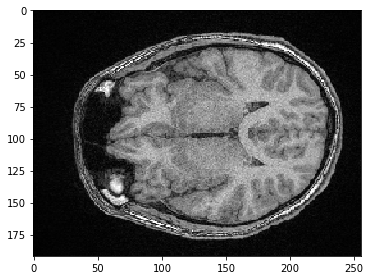
Um 3D-Bilder mit scikit-image’s imshow zu visualisieren, müssen wir einen Schnitt zur Visualisierung auswählen. Zum Beispiel einen Z-Schnitt:
slice_image = image[100]
imshow(slice_image)
<matplotlib.image.AxesImage at 0x2b54f73d340>
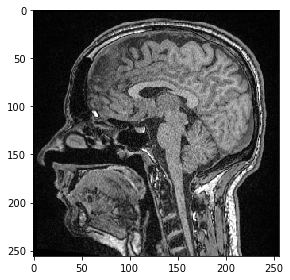
Wir können auch eine Ebene auswählen, bei der alle Pixel die gleiche Y-Position haben. Wir müssen nur angeben, dass wir alle Pixel in Z mit der : Syntax behalten möchten.
slice_image = image[:, 100]
imshow(slice_image)
<matplotlib.image.AxesImage at 0x2b54f836af0>
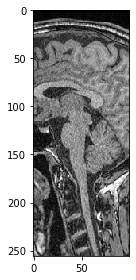
Zuschneiden#
Wir können auch einen Teilstapel mit Hilfe von Indexierung in den eckigen Klammern auswählen.
sub_stack = image[50:150]
sub_stack.shape
(100, 256, 256)
Wir können auch eine Teilregion in X auswählen. Wenn wir alle Pixel entlang Z und Y (die ersten beiden Dimensionen) behalten wollen, geben wir einfach : an, um alle zu behalten.
sub_region_x = image[:, :, 100:200]
imshow(sub_region_x[100])
<matplotlib.image.AxesImage at 0x2b54f8ae850>
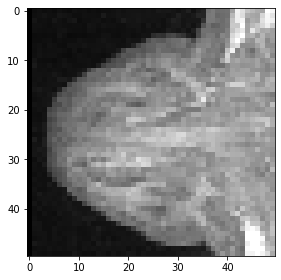
Um alle Pixel in einer Richtung oberhalb eines bestimmten Wertes auszuwählen, müssen wir nur den Start vor : angeben.
sub_region_y = image[:, 100:]
imshow(sub_region_y[100])
<matplotlib.image.AxesImage at 0x2b54f90ae20>
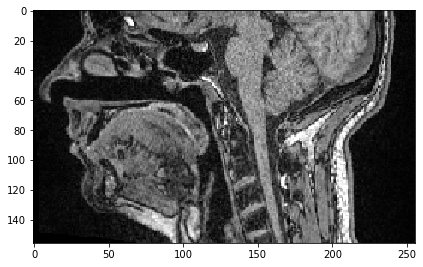
Ähnlich können wir alle Pixel bis zu einer bestimmten Position auswählen.
sub_region_x2 = image[:, :, :50]
imshow(sub_region_x2[100])
<matplotlib.image.AxesImage at 0x2b550943d30>
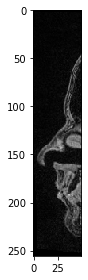
Zu guter Letzt, so wird ein zugeschnittener Würfel spezifiziert.
cropped_cube = image[80:130, 120:170, :50]
cropped_cube.shape
(50, 50, 50)
imshow(cropped_cube[20])
<matplotlib.image.AxesImage at 0x2b5509a4a00>
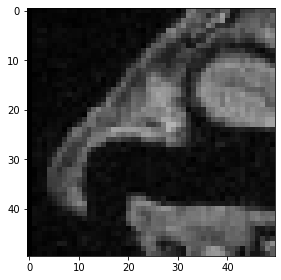
Und so sehen Maximumintensitätsprojektionen dieses zugeschnittenen Würfels aus.
maximum_intensity_projection_along_z = np.max(cropped_cube, axis=0)
imshow(maximum_intensity_projection_along_z)
<matplotlib.image.AxesImage at 0x2b550a03f40>
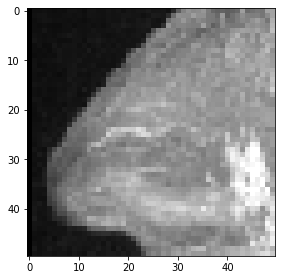
maximum_intensity_projection_along_y = np.max(cropped_cube, axis=1)
imshow(maximum_intensity_projection_along_y)
<matplotlib.image.AxesImage at 0x2b550a5e8e0>