Benachbarte Zellen zählen#
In diesem Notebook visualisieren wir, welche Zellen in einem Gewebe benachbart sind, indem wir ein Netz zeichnen. Darüber hinaus können wir die Intensität des Netzes so ändern, dass sie dem Abstand zwischen den Schwerpunkten der Zellen entspricht.
import pyclesperanto_prototype as cle
import numpy as np
import pandas as pd
Testdaten#
Lassen Sie uns eine gewebeähnliche Struktur erzeugen, die aus Zellen besteht, die typischerweise etwa 6 Nachbarn haben.
cells = cle.artificial_tissue_2d(
delta_x=48,
delta_y=32,
random_sigma_x=7,
random_sigma_y=7,
width=250,
height=250)
cle.imshow(cells, labels=True)

Netz zwischen benachbarten Zellen#
Bevor wir Nachbarn zählen, sollten wir die Nachbarschaftsbeziehungen visualisieren. Wir können dies tun, indem wir ein Netz zwischen den Schwerpunkten benachbarter Zellen zeichnen.
mesh = cle.draw_mesh_between_touching_labels(cells)
cle.imshow(mesh)

Wir können auch beide Visualisierungen in einem Bild kombinieren. Beachten Sie, dass diese Bilder nicht für weitere quantitative Analysen verwendet werden sollten. Sie dienen nur der Visualisierung.
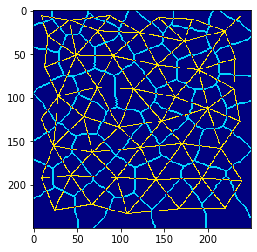
Schwerpunktverbindungen und Zellgrenzen#
Eine gängige Methode zur Visualisierung von Geweben in diesem Zusammenhang besteht darin, Zellgrenzen und das Schwerpunktnetz in verschiedenen Farben zu zeichnen.
visualization = mesh * 2 + cle.detect_label_edges(cells)
cle.imshow(visualization, color_map='jet')
Analyse und Visualisierung der Anzahl benachbarter Zellen#
Wir können auch die benachbarten Zellen zählen und das Ergebnis als parametrisches Bild in Farben visualisieren.
neighbor_count_image = cle.touching_neighbor_count_map(cells)
cle.imshow(neighbor_count_image, color_map='jet', colorbar=True, min_display_intensity=0)

Beachten Sie, dass die Zahlen entlang des Bildrandes möglicherweise nicht genau sind. Daher sollten wir die entsprechenden Zellen von der weiteren Analyse ausschließen.
cells_ex_border = cle.exclude_labels_on_edges(cells)
cle.imshow(cells_ex_border, labels=True)

Nach der Korrektur des Labelbildes können wir auch das parametrische Bild korrigieren.
neighbor_count_image_ex_border = neighbor_count_image * (cells_ex_border != 0)
cle.imshow(neighbor_count_image_ex_border, color_map='jet', colorbar=True, min_display_intensity=0)

Jetzt können wir die Anzahl der Nachbarn messen. Wir können entweder einfach diese Zahlen lesen und sie in eine Liste einfügen …
cle.read_intensities_from_map(cells_ex_border, neighbor_count_image_ex_border)
cl.Array([[0., 5., 6., 7., 6., 6., 7., 6., 6., 5., 5., 6., 6., 7., 6., 6.,
6., 6., 6., 6., 6.]], dtype=float32)
… oder wir können diese Werte zusammen mit allen anderen Statistiken lesen und sie in einen pandas DataFrame einfügen.
statistics = cle.statistics_of_labelled_pixels(neighbor_count_image_ex_border, cells_ex_border)
table = pd.DataFrame(statistics)
# Spalte umbenennen
table = table.rename(columns={"mean_intensity": "number_of_neighbors"})
# Nur eine Teilmenge aller Spalten herausfiltern; nur das, was uns interessiert
table = table[["label", "number_of_neighbors", "centroid_x", "centroid_y"]]
table
| label | number_of_neighbors | centroid_x | centroid_y | |
|---|---|---|---|---|
| 0 | 1 | 5.0 | 74.592697 | 23.731028 |
| 1 | 2 | 6.0 | 173.741379 | 35.582230 |
| 2 | 3 | 7.0 | 49.460915 | 51.928082 |
| 3 | 4 | 6.0 | 91.783768 | 52.666199 |
| 4 | 5 | 6.0 | 142.728210 | 62.962471 |
| 5 | 6 | 7.0 | 189.973206 | 68.526794 |
| 6 | 7 | 6.0 | 73.699181 | 88.797356 |
| 7 | 8 | 6.0 | 115.321083 | 90.665649 |
| 8 | 9 | 5.0 | 163.097733 | 99.824242 |
| 9 | 10 | 5.0 | 58.206852 | 122.341309 |
| 10 | 11 | 6.0 | 98.847565 | 134.280228 |
| 11 | 12 | 6.0 | 147.731705 | 127.326218 |
| 12 | 13 | 7.0 | 200.522903 | 112.817368 |
| 13 | 14 | 6.0 | 67.880905 | 162.991592 |
| 14 | 15 | 6.0 | 122.943306 | 157.515717 |
| 15 | 16 | 6.0 | 171.010025 | 153.333328 |
| 16 | 17 | 6.0 | 43.426266 | 189.492981 |
| 17 | 18 | 6.0 | 96.272728 | 192.326477 |
| 18 | 19 | 6.0 | 140.815964 | 194.446030 |
| 19 | 20 | 6.0 | 193.817596 | 193.019318 |
Übung#
Analysieren Sie ein größeres Sichtfeld mit mehr Zellen und variieren Sie die Parameter random_sigma_x und random_sigma_y der Funktion artificial_tissue_2d. Verwenden Sie eine Karte der Berührungsnachbarnzahl, um die Anzahl der sich berührenden Nachbarn vor und nach der Anwendung eines Medianfilters auf die Karte zu zählen.
