Nachbearbeitung für membranbasierte Zellsegmentierung#
In diesem Notebook verwenden wir einen Seeded-Watershed-Algorithmus von napari-segment-blobs-and-things-with-membranes, um Zellen zu segmentieren und anschließend zu filtern, um fälschlicherweise segmentierte Objekte zu entfernen.
import matplotlib.pyplot as plt
import numpy as np
from skimage.io import imread
import pyclesperanto_prototype as cle
import napari_segment_blobs_and_things_with_membranes as nsbatwm
Die folgenden Beispielbilddaten sind eine Zeitreihe von Zellen, die mit einem Membranmarker markiert sind. Der Datensatz ist eine Zurverfügungstellung von Sascha M. Kuhn, Nadler Lab, MPI-CBG Dresden.
image_timelapse = imread("../../data/membrane_2d_timelapse.tif")
image_timelapse.shape
(5, 256, 256)
Wir beginnen damit, ein einzelnes Kanal-, Einzelzeitpunkt-Beispiel aus der Mitte des Zeitreihen-Stacks zu extrahieren.
membranes_single_slice = image_timelapse[2]
membranes_single_slice.shape
(256, 256)
cle.imshow(membranes_single_slice)
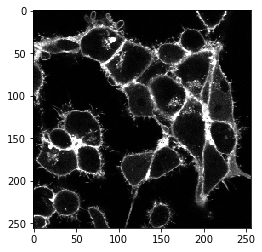
Das napari-segment-blobs-and-things-with-membranes Plugin ist skriptfähig. Alle Befehle, die aus dem Menü aufgerufen werden können, können auch von Python aus aufgerufen werden. Zum Beispiel kann der lokale-Minima-Seeded-Watershed wie folgt aufgerufen werden:
cell_labels_pre = nsbatwm.local_minima_seeded_watershed(membranes_single_slice, spot_sigma=7)
cle.imshow(cell_labels_pre, labels=True)

Offensichtlich werden nach diesem Schritt zu viele Objekte segmentiert und markiert. Daher sollten wir untersuchen, welche Eigenschaften dieser Zellen es uns ermöglichen, echte Zellen und segmentierten Hintergrund zu unterscheiden. Da die Zellen beispielsweise auch vorne und hinten Membran haben, nehmen wir dieses Signal als höhere Intensität in echten Zellen wahr. Um dies quantitativ zu visualisieren, erstellen wir eine Karte der mittleren Intensität der Zellen. In dieser Karte erhält jeder Pixel, der zu einer bestimmten Zelle gehört, die durchschnittliche Signalintensität der gesamten Zelle.
mean_intensity_map = cle.mean_intensity_map(membranes_single_slice, cell_labels_pre)
cle.imshow(mean_intensity_map, colorbar=True, colormap='jet')
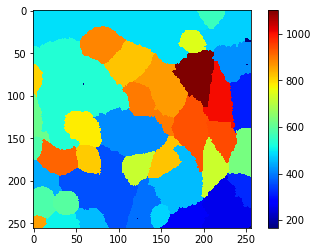
Aus dieser Ansicht und der Farbskala an der Seite können wir einen Schwellenwert schätzen, z.B. 650, der es uns ermöglicht, echte Zellen vom Hintergrund zu trennen. Wenn eine solche einfache Schwellenwertmethode nicht funktioniert und/oder ein einzelner Parameter wie die mittlere Intensität diese Unterscheidung nicht zulässt, kann es sinnvoll sein, Objektklassifikatoren, auch bekannt als maschinelles Lernen, zu verwenden. Zum Beispiel können das napari-accelerate-pixel-and-object-classification Plugin und die zugrunde liegende apoc-Bibliothek helfen.
cell_labels = cle.exclude_labels_with_map_values_within_range(mean_intensity_map, cell_labels_pre, maximum_value_range=700)
cle.imshow(cell_labels, labels=True)

Anwendung des Workflows auf die gesamte Zeitreihe#
Wir haben jetzt eine grobe Vorstellung davon, wie ein Workflow zur Segmentierung der Zellen aussieht. Wir schreiben den gesamten Workflow in eine Funktion um. Die Funktion hat auch eine ordentliche Dokumentation, damit das zukünftige Ich noch weiß, was die Funktion tut.
def cell_segmentation_worflow(membranes_single_slice, cell_segmentation_spot_sigma=7, maximum_mean_intensity_per_cell=700):
"""Zellsegmentierung basierend auf Membransignal
Parameter
----------
membranes_single_slice
Zu segmentierendes Bild
cell_segmentation_spot_sigma: float, optional
Ermöglicht die Konfiguration der Segmentierung. Je höher Sigma, desto weniger Zellen werden vorhanden sein.
maximum_mean_intensity_per_cell: float, optional
Schwellenwert für die mittlere Intensität pro Zelle. Zellen mit Signal darunter werden ausgeschlossen
Rückgabe
-------
Zell-Label-Bild
"""
# Zellkandidaten markieren
cell_labels_pre = nsbatwm.local_minima_seeded_watershed(
membranes_single_slice,
spot_sigma=cell_segmentation_spot_sigma)
# Intensität in allen Kandidaten messen
mean_intensity_map = cle.mean_intensity_map(
membranes_single_slice,
cell_labels_pre)
# Kandidaten mit niedrigem Signal ausschließen
cell_labels = cle.exclude_labels_with_map_values_within_range(
mean_intensity_map, cell_labels_pre,
maximum_value_range=maximum_mean_intensity_per_cell)
return cell_labels
Diese Funktion können wir auf die gesamte Zeitreihe anwenden und überprüfen, ob die Segmentierung auch in allen Zeitpunkten gut funktioniert.
for t in range(len(image_timelapse)):
# einzelne Scheibe / Zeitpunkt extrahieren
membranes_single_slice = image_timelapse[t]
# Zellen segmentieren
cell_labels = cell_segmentation_worflow(membranes_single_slice)
# Überschrift ausgeben
print("t = ", t, ", Anzahl der Zellen: ", cell_labels.max())
# drei Bilder anzeigen: Bild, Labels, Überlagerung
fix, axs = plt.subplots(1, 3, figsize=(10,10))
cle.imshow(membranes_single_slice, plot=axs[0])
cle.imshow(cell_labels, plot=axs[1], labels=True)
cle.imshow(membranes_single_slice, plot=axs[2], alpha=0.5, continue_drawing=True)
cle.imshow(cell_labels, plot=axs[2], labels=True, alpha=0.5)
plt.show()
t = 0 , number of cells: 17.0
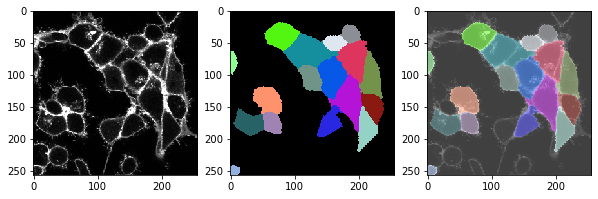
t = 1 , number of cells: 18.0

t = 2 , number of cells: 17.0
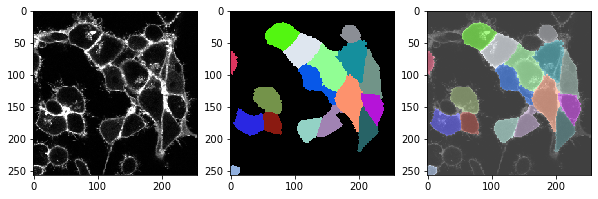
t = 3 , number of cells: 18.0
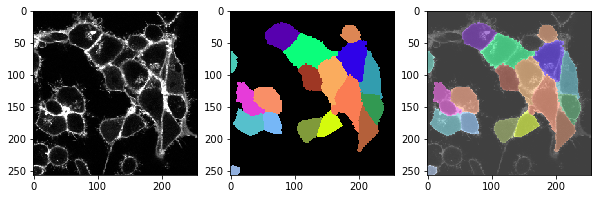
t = 4 , number of cells: 17.0
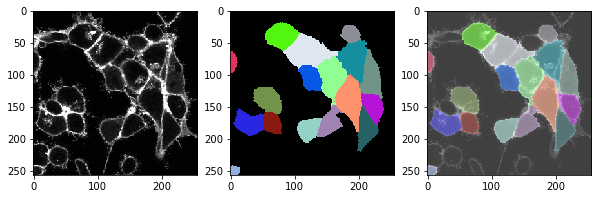
Übung#
Anstatt Zellen mit niedriger Signalintensität herauszufiltern, filtern Sie Zellen heraus, die groß sind und/oder den Bildrand berühren. Wenden Sie den neuen Workflow auf die gesamte Zeitreihe an.
